限制酶的種類和命名
限制酶的命名是以該酶來源的原核生物的名稱爲依據的,即酶的名稱的第一個大寫字母取自於屬名的第一個字母,第二、三個小寫字母取自於種名的前兩個字母,字母後面的羅馬字則是簡單地表明該種生物中不同限制酶分離的先後順序。在正式的出版物中,限制酶名稱的前三個字母常用斜體或加下橫線表示。例如, 分離自産色鏈黴菌(Streptomyces achromogenes )的兩種限制酶被分別命名爲SacI和 SacII。如果同一生物種內又分爲不同的血清型或菌株,其名稱則放在限制酶名稱的第三個字母之後。比如限制酶HincⅡ和HindⅢ則是分別來自流感嗜血菌(Haemophilus
influenzae)的c和d血清型菌株。
迄今爲止,已有上千種限制酶被分離純化,這些酶幾乎全是從真細菌(eubacteria )和古細菌(archaeobacteria )中分離得到的。從目前已商品化的100多種限制酶的識別序列可以看出,限制酶所識別的DNA序列大多數都富含胞嘧啶(C)和鳥嘌呤(G)(6.7)。
限制性內切酶的識別序列與酶切特點
由於DNA分子是由四種核苷酸組成的,那麽它們就會有不同的組合方式。不同長短的核苷酸序列就會顯現出一些特殊的組合。比如由4種核苷酸組成的4核苷酸序列可以有24種排列方式,其中有8種4核苷酸具有特殊的結構:
1. 5’-CATG-3’
2. 5’-CTAG-3’
3. 5’-GTAC-3’
4. 5’-GATC-3’ 3’-GTAC-5 3’-GATC-5’ 3’-CATG-5’ 3’-CTAG-5’
5. 5’-ACGT-3’
6. 5’-AGCT-3’
7. 5’-TGCA-3’
8. 5’-TCGA-3’3’-TGCA-5’ 3’-TCGA-5’ 3’-ACGT-5’ 3’-AGCT-5’
這8種核苷酸序列都有一個共同的特點,那就是無論從那一條鏈按5’à 3’方向讀這些序列,兩條鏈上的序列都是相同的。比如第4個序列,上面一條鏈按5’à 3’方向是GATC,下面一條鏈按5’à 3’方向讀序列仍是GATC。這就是DNA的回紋結構。限制酶識別的DNA特異序列就是這種具有回紋結構的序列,我們常常就稱之爲限制酶識別序列或識別位點。限制酶識別序列可以有多長呢?現在已從各種微生物中發現上千種限制酶,它們識別序列的長度最短的是4個核苷酸,就如上面所例舉的例子,最長的爲8個核苷酸。基因clone過程中使用頻率最高的是識別6個核苷酸的限制酶。這是爲什麽呢?因爲由4個核苷酸組成的識別序列在DNA分子中出現的頻率很高,如果按完全隨機分佈的原則,每44=256個核苷酸可出現一個相同的4核苷酸識別序列。如果是由6個核苷酸序列組成的限制酶識別位點,那麽就應該有46=4096 bp才可能出現一次。識別8個核苷酸的限制酶識別位點就應該有48=65,536 bp才重復一次。因此,在一個DNA分子中,識別4個核苷酸的限制酶位點太多,識別8核苷酸的限制酶位點又太少。換句話說,識別4核苷酸的限制酶將DNA切得太短,識別8個核苷酸的限制酶將DNA切得太長,而識別6個核苷酸的限制酶則比較適中,切下的DNA片段平均長4.1 kb左右。除此之外,片段過長或過短,從技術角度講,操作起來也不太方便。實際上,任何一種生物基因組的DNA分子中的核苷酸分佈實際上並不是完全隨機的。比如,在真核生物的DNA分子中,2核苷酸序列CG是很少見的,所以在SV40(Simian vacuolating virus,猿猴空泡病毒)的DNA分子中(基因組長爲5,243 bp)僅有一個HpaII位點(識別序列爲 C↓CGG),而ThaI位點(識別序列爲CG↓CG)則根本沒有。 但是,在大小相似的原核生物DNA分子中,如最常見的大腸桿菌質粒載體pBR322(總長4,363 bp)則有26個HpaII位點和23個ThaI位點。在一個含有75% AT和25% GC的DNA分子中,每262,144
bp則只有一個SmaI位點(識別序列CCC↓GGG)。實際上,限制酶SmaI不能將一種Euglena gracilis的葉綠體DNA分子(130,000 bp)切爲兩段,這表明該分子中沒有SmaI位點。這些資料對於限制酶的選擇是十分有用的。比如在建立基因組文庫時,DNA片段要求盡可能長一些以便使這些片段含有完整的基因,這就需要選擇一些限制酶,它們的識別序列在該生物DNA 分子中應當是一些不常見的核苷酸序列,而在繪製限制酶圖譜時, 選擇的限制酶則應該識別較爲常見的識別序列。 當一個DNA分子被限制酶切開後, 切點處必然會産生兩個末端。 這就像用剪刀把一根繩子剪成兩段時的情形一樣。由於DNA分子是由兩條互補鏈構成,因而酶切後的兩個新末端都有一個5’-端和3’-端,此時兩個末端雙鏈中的5'端是PO4基, 而3' 端則是OH基。不同的限制酶産生的末端結構可分爲三類:平端, 5'-端突起和3'-端突起, 但每種限制酶則只能産生一種類型的末端。凡是識別奇數個核苷酸(5個或7個)的限制酶只能産生突起末端, 而識別偶數個核苷酸(4個、6個或8個)的限制酶則可能産生平端或突起末端。如果切點是在中軸位置時, 産生的末端是平端, 如PuvⅡ:
5'──CAG↓CTG──3' PvuII
5'──CAGOH
PCTG──3'
3'──GTC↑GAC──5' ────→ 3'──GTCP
OHGAC──5'
如果切點在中軸的左上側和右下側, 則産生5'-端突起;如果切點在中軸的左上側和右下側, 則産生5'-端突起。如EcoRI和PstI酶切結果:
5'──G↓AATTC──3' EcoRI
5'──GOH
PAATTC──3'
3'──CTTAA↑G──5'────→ 3'──CTTAAP
OHG──5'
5'──CTCGA↓G──3' PstI
5'──CTCGAOH
PG──3'
3'──G↑AGCTC──5' ───→ 3'──GP
OHAGCTC──5'
分子clone中所用的限制酶産生的突起末端的單鏈部分含有的核苷酸數目爲可1-5個,但多數爲2個和4個。如上述的EcoRI和PstI酶切後的DNA都是産生4個核苷酸突起,産生2個、3個和5個核苷酸突起的限制酶分別舉例如下:
5' ─A↓TCGAT─ 3' ClalI
5' ─ATOH PCGAT─ 3'
3’ ─TAGCT↑A─ 5' ───→ 3' ─TAGCP HOTA─ 5'
5' ─G↓GNCC─ 3' Sau96I
5'─GOH PGNCC─ 3'
3' ─CCNG↑G─ 5' ───→ 3'─CCNGP
HOG─ 5'
5' ─G↓GTNACC─ 3' BstEⅡ 5' ─GOH PGTNACC─ 3'
3' ─CCANTG↑G─ 5' ────→ 3' ─CCANTGP HOG─ 5'
在多數情況下,同一種限制酶所産生的突起末端結構總是相同的,因而用同一種限制酶切的同個或兩個不同來源的DNA分子所産生的末端都可以相互配對, 在DNA連接酶的作用下,磷酸二酯鍵可被修復而成爲一個重組的DNA分子。凡是那些識別序列是唯一的限制酶都屬於這一類。由於這些限制酶的識別序列都是對稱的,因而兩個DNA片段可以從兩個不同的方向連接起來。這種外源DNA片段的插入方向有時並不顯得重要,如在基因組文庫構建時。然而在研究基因的表達時,外源DNA 的插入方向則是十分重要的。 雖然大多數同種限制酶産生的末端是相同的,但是有的同種限制酶産生的末端則是不同的。因而即使利用同種限制酶作用DNA 分子,各切點處産生的末端結構可能就不一定相同。正是因爲末端結構不相同,也就無法將它們互相連接起來。凡是那些可識別簡並序列的限制酶都屬於這一類,如限制酶AvaI能識別四種6核苷酸序列。顯然, 當用AvaI在兩個識別位點處將DNA切開後, 它們能互相連接起來的可能性僅爲1/4:
5' ─C CCGGG─ 3' 5'─C CCGAG─ 3'
3' ─GGGCC C─ 5' 3'─GGGCT C─ 5'
5' ─C TCGGG─ 3' 5'─C TCGAG─ 3'
3' ─GAGCC C─ 5' 3'─GAGCT C─ 5'
這相同切點的限制酶4種末端中的任意一種是無法與其他3種末端相連。
雖然不同種的限制酶産生的突起末端在大多數情況下是不相同的,但有的不同種限制酶産生的末端卻是相同的,即具有相同類型的突起末端(都是5'-端或3'-端突起),突起的核苷酸數目相等且序列也相同,因而可以互相連接起來。這種由不同種的限制酶産生的能相互連接的末端常稱之爲相容性末端,這些酶則稱之爲同尾酶。例如,限制酶BamHI,BalI,BglⅡ和XhoI都能識別各自的6核苷酸序列,且切點都在同一位置,依次爲GGATCC,TGATCA,AGATCT和 PuGATCPy, 因此産生的DNA片段都産生一個相同的單鍵5'-端突起(突起序列是GATC)。當這些酶作用DNA分子後,它們都能相互連接,但是新形成的DNA分子將同時失去這兩種酶的識別序列。例如:
5'──A↓TATCT──3' BglII
5'──A GATCT──3'(1)
3'──TCTAG↑C──5' ──→ 3'──TCTAG A──5' (2)
5'──G↓GATCC──3' BamHI
5'──G GATCC──3'(3)
3'──CCTAG↑G──5' ──→ 3'──CCTAG G──5'(4)
(1)和(4)末端連接得:
5'──AGATCC──3'
3'──TCTAGG──5'
(2)和(3)末端連接得:
5'──GGATCT──3'
3'──CCTAGA──5'
Restriction
enzyme先在雙股DNA上找到其recognition site,再從recognition site找到cleavage site,並進行endonuclease的酵素作用,將cleavage site的phosphodiester bond水解,recognition site及cleavage site並不一定在同一個位置上,type Ⅰ限制酵素的cleavage site在離recognition site約400~7000bp以外的任何一個位置上﹔type Ⅱ限制酵素的cleavage site在recognition site內或鄰近的特定位置上﹔type Ⅲ限制酵素的cleavage site在離recognition site約25~27bp以外的任何或特定位置上。限制酵素在辨認的DNA序列中間,對雙股DNA同時進行切割稱blunt end。如SmaⅠ
5’CCCGGGOH3’ => 5’CCCOH3’ + 5’GGG3’
3’GGGCCCP5’
3’GGGP5’ 3’CCC5’
限制酵素切再所辨認的二股DNA序列上的位置並不同,二切點會相個數個核甘酸,而留下長短DNA稱為sticky end。依兩切點的位置不同,可分為3’cohesive end和5’ cohesive end。
3’cohesive end如PstⅠ
5’CTGCAG3’ => 5’CTGCAOH3’ + 5’G3’
3’GACGTC5’
3’GP5’
3’ACGTC5’
5’ cohesive end如EcoRI
5’GAATTC3’ => 5’GOH3’
+ 5’AATTC3’
3’CTTAAG5’
3’CTTAAP5’
3’G5’
有一些不同限制酵素辨認相同DNA序列的例子,這些限制酵素則稱為isoschizomers。
限制酵素辨認相同DNA序列,且切割在相同位置者稱perfect isoschizomers。
如HindⅢ和HsuⅡ均辨認
5’AAGCTTOH3’ =>
5’AOH3’ + 5’AGCTT3’
3’TTCGAAP5’ 3’TTCGAP3’ 3’A5’
限制酵素辨認相同DNA序列,但切割在不同的位置者稱為imperfect isoschizomers。
如SmaⅠ和XmaⅠ
SmaⅠ
5’CCCGGGOH3’ => 5’CCCOH3’ + 5’GGG3’
3’GGGCCCP5’
3’GGGP5’ 3’CCC5’
XmaⅠ
5’CCCGGGOH3’ => 5’COH3’ + 5’CCGGG3’
3’GGGCCCP5’
3’GGGCCP5’ 3’C5’
限制酶使用時buffer的選擇
buffer中含Tris-HCl、MgCl2、NaCl、KCl、DTT(為抗氧化劑,可穩定含sulfhydryl groups的酵素及蛋白質)。buffer提供限制酵素作用最佳的pH值及離子濃度,不同的限制酵素的最佳作用條件並不相同,故要使用不同的buffer,且進行限制酵素處理時,若使用buffer不當,會有star activity,而star activity是指限制酵素對所作用的DNA及序列失去專一性,當酵素辨認切割位置的能力降低,導致相似的序列或是錯誤的辨認序列長度也會作用,而產生錯誤的結果。所以當DNA同時以兩種限制酵素處理時,選擇可使兩種酵素之活性反應均可達75﹪以上的restriction buffer,若找不到適當的buffer則先加低鹽的buffer使其中一酵素作用後,在加入高鹽buffer使另一酵素作用,若無法以鹽的高低分別加入不同的buffer,則先加入一種buffer作用後,加3M NaOAc/95﹪alcohol沉澱DNA,再加另一種buffer作用。
Buffer提供限制酵素作用最佳的pH值即離子濃度,不同的限制酵素的最佳作用條件並不相同,因此需使用不同種類的buffer。
不同廠商出產的buffer 成分略有不同,在此以Promega所出的buffer為例
限制酵素buffer的組成(composition) (1X)
|
Buffer
|
pH(37℃)
|
Tris-HCl(mM)
|
MgCl2(mM)
|
NaCl(mM)
|
KCl(mM)
|
DTT(mM)
|
|
A
|
7.5
|
6
|
6
|
6
|
-
|
1
|
|
B
|
7.5
|
6
|
6
|
50
|
-
|
1
|
|
C
|
7.9
|
10
|
10
|
50
|
-
|
1
|
|
D
|
7.9
|
6
|
6
|
150
|
-
|
1
|
|
E
|
7.5
|
6
|
6
|
100
|
-
|
1
|
|
G
|
8.2
|
50
|
5
|
-
|
-
|
-
|
|
H
|
7.5
|
90
|
10
|
50
|
-
|
-
|
限制酵素在Promega’s 10X buffer的相對活性(relative activity)
|
限制
|
附屬
|
酵素活性
|
作用溫度
|
|
酵素
|
buffer
|
A
|
B
|
C
|
D
|
M
|
|
|
Bam HI
|
E
|
75-100﹪
|
75-100﹪
|
75-100﹪
|
50-75﹪
|
75-100﹪
|
37℃
|
|
Eco RI
|
H
|
25-50﹪
|
50-75﹪
|
50-75﹪
|
50-75﹪
|
100﹪
|
38℃
|
|
Hind III
|
E
|
25-50﹪
|
100﹪
|
75-100﹪
|
50-75﹪
|
50-75﹪
|
39℃
|
|
Pst I
|
H
|
10-25﹪
|
50-75﹪
|
50-75﹪
|
50-75﹪
|
25-50﹪
|
40℃
|
限制酶在基因工程上的利用
根據基因工程的概念,基因工程(亦稱分子clone或DNA 重組技術)應是在分子水平上將目標基因或具有其他功能的DNA片段按人們的要求和設計,在體外重新組合成具有某種新的功能或比原DNA序列更爲有效的重組DNA分子的過程,然後利用合適的方法將重組DNA分子導入劃或轉移到感興趣的細胞或生物體內,新的性狀就有可能産生,新的遺傳材料也就形成了。這種新材料常稱之爲轉基因生物。很顯然,在DNA的重組過程中,需要將原來生物的DNA(稱之爲供體DNA)切斷,然後與一種稱之爲clone載體的DNA分子連接起來,再通過適當方法轉移到某種宿主細胞(亦稱受體細胞)中進行DNA擴增或基因表達。因此,在切割DNA和連接DNA的過程中需要利用各種生物催化劑—酶,我們常常把應用於基因工程研究中使用的酶稱之爲基因工程工具酶,或分子clone工具酶。分子clone工具酶是一類用於DNA重組過程中不同DNA分子的製備、切割、修飾、擴增, 核酸分子的標記以及它們的核苷酸序列測定的酶。大多數分子clone工具酶都是來自不同的微生物和噬菌體, 有少數酶則是來自動物和動物病毒。由於基因工程技術的迅猛發展, 一些爲工具酶編碼的基因也被clone出來, 並用大腸桿菌細胞來生産這些工具酶。限制性內切酶廣泛地應用于各種DNA分子的體外重組,限制酶譜的繪製,基因的次clone分析,基因和染色體結構的分析以及基因的體外突變等諸方面。
ensonuclease的功用
雖然限制酶反應本身是十分簡單的,但在限制酶的選擇上往往極富挑戰性,有時會因爲一個限制酶切位點選擇不當而事倍功半,甚至根本無法達到預期目標。應付這種挑戰性的基礎很簡單, 就是要認真瞭解不同的限制酶有不同的識別序列和酶切特徵。由於以後的章節還會多次涉及限制酶的用途,因而在此不再贅述。
T4 DNA連接酶
T4 DNA連接酶(T4 DNA Ligase)是分子clone實驗中最常用的工具酶之一,該酶催化雙鏈DNA 分子中相鄰的3'-羥基和5'-磷酸基間磷酸二酯鍵的形成,它既可以連接兩個具有粘性末端的 DNA片段,也可以連接兩個具有平端的DNA片段,但是連接後者所需的酶量往往是連接前者的50倍。
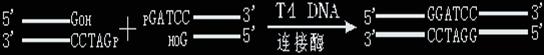
具有平端的DNA分子間的連接作用在分子clone實驗中有著廣泛的應用。 這是因爲該連接反應不需要DNA末端具有粘性,因而任何具有平端的DNA片段均可連接起來。這對於那些不具有合適的限制酶位點的DNA片段尤爲重要。此外, 利用超聲波或DNaseI降解所獲得的染色體DNA隨機片段也可以clone到載體DNA中的平端位點。在分子clone過程中,有時爲了避免載體DNA的自我連接(Self-ligation), 常常要將限制酶所産生的5'-磷酸基用磷酸酶(見後述)除去,因而當用連接酶將載體DNA和外源DNA分子連接時,DNA雙鏈中只有一條鏈是連接起來的,另一條鏈的連接則是把DNA轉移到宿主細胞後由細胞內的連接酶完成的(7)。
DNA聚合酶
在基因工程操作中,常常需要以DNA或RNA作爲模板合成新的DNA鏈,這些新合成的鏈或是用於DNA標記,或是用於DNA序列分析,或是用於DNA重組,或是用於某些感興趣的DNA片段的擴增。 因此,不同的DNA 聚合酶或反轉錄酶可用於上述的不同目的。
大腸桿菌DNA聚合酶I
從大腸桿菌中已分離純化出三種DNA聚合酶(DNA Polymerase,常簡寫爲Pol), 即PolI,PolⅡ和PolⅢ。PolI和PolⅡ主要參與DNA的修復過程,而PolⅢ則參與DNA的複製過程,只有PolI廣泛用於分子clone。大腸桿菌聚合酶I在有引物和脫氧核苷酸存在時,能以單鏈DNA作爲模板和以5'→3'的方向合成一條新鏈。除聚合酶活性外,PolI 還具有3'→5'和5'→3'的外切酶活性。所謂外切酶活性就是從DNA分子鏈的末端開始,一個或幾個核苷酸被逐漸切除的過程。這兩種外切酶活性不僅在DNA末端的選擇性和切割的方向上存在著差異,而且其他一些基本特徵也不相同。如5'→3'外切酶只裂解雙鏈區域內的磷酸二酯鏈,而3'→5'外切酶則只作用于單鏈DNA。
大腸桿菌PolI在蛋白酶的作用下可被切爲兩段,其中分子量較大的一段常稱之爲DNA聚合酶大片段,或稱Klenow片段,寫作polIK,它仍具有DNA聚合酶活性和3'→5'的外切酶活性。DNA聚合酶I的主要用途是標記DNA鏈,即切口移位(nick-translation),polIK則主要用於DNA鏈的合成,包括補平DNA末端、DNA測序等反應。(7)

圖 DNA聚合酶的5’® 3’外切酶活性和聚合作用
1. http://www.nobel.se/medicine/laureates/1978/index.html
2. http://www.nobel.se/medicine/laureates/1978/arber-autobio.htm
3. http://www.nobel.se/medicine/laureates/1978/nathans-autobio.html
4. http://www.nobel.se/medicine/laureates/1978/smith-autobio.html
5. http://www.biol.ntnu.edu.tw/NSC/bionetwork/biorelative/scissors.html
6.http://bioteach.ttu.edu.tw/highschool/restrict.htm
7. http://www.fuxing.sh.cn/stuweb/g199803/basic/phnase.htm