ExPASy資料庫簡介-蛋白質序列分析與預測(3)
口述:呂平江 老師
整理:林崇文、鄭詩思、李佩真

![]()
![]()



![]()

![]()
![]()

![]()

![]()
![]()


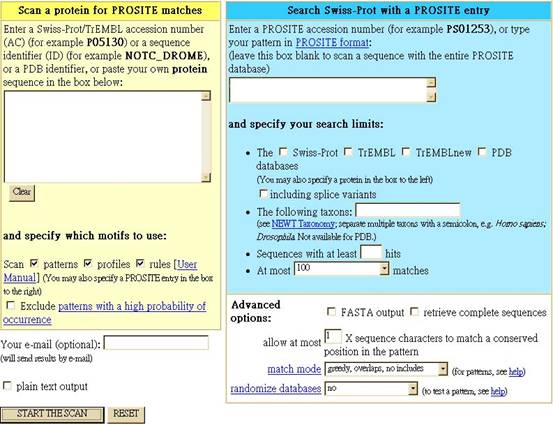
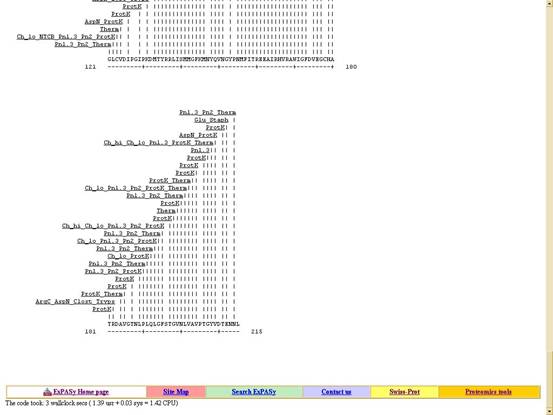
在要進行protein的比對之前,可以利用ScanProsite先進行一些相關的比對,找出一些可能的modification site。利用2003/05/01公佈在NCBI上的SARS coronavirus Hong Kong/03/2003 RNA-directed RNA polymerase gene來做試驗。
![]()
![]()


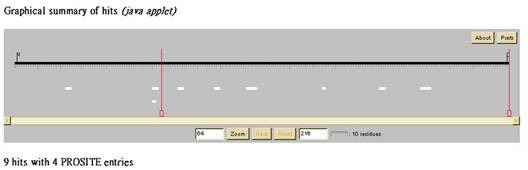

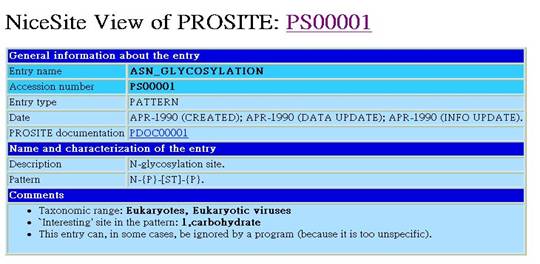 若是要進一步了解各partten的特性,可以點選在java
applet圖中的白色區域,會秀出較詳細的資訊。例如:87-90
Asparagine glycosylation的區域→
若是要進一步了解各partten的特性,可以點選在java
applet圖中的白色區域,會秀出較詳細的資訊。例如:87-90
Asparagine glycosylation的區域→

![]()
![]()



![]()
![]()




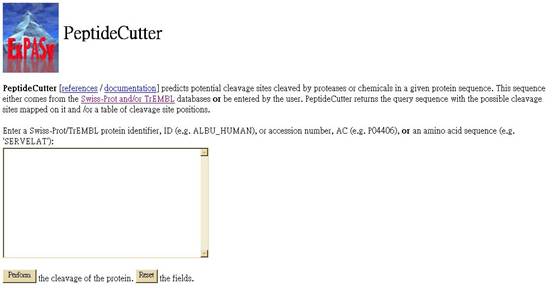
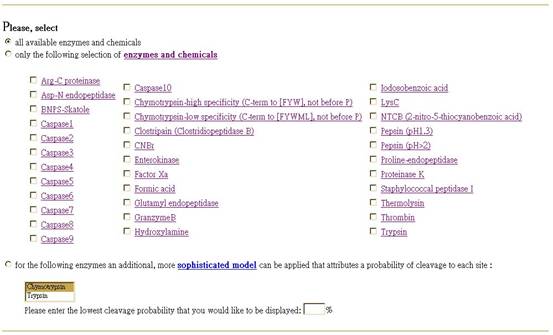
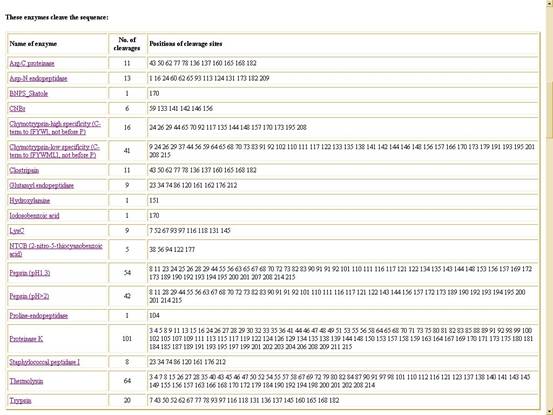
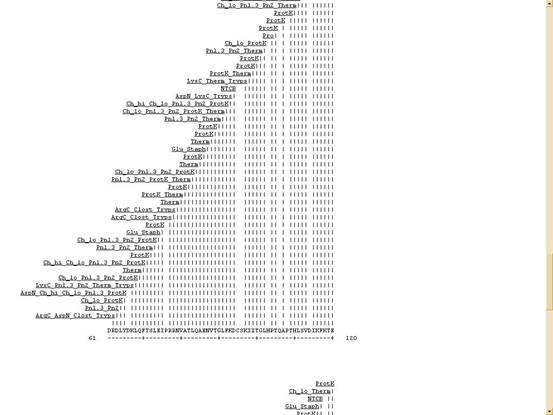
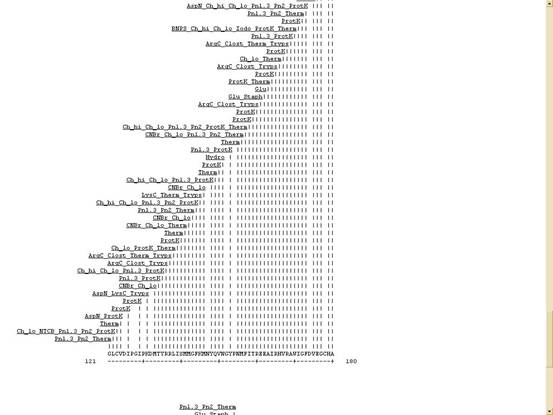
![]() 利用電腦來模擬用restriction
enzyme作用在peptide下的結果。例如:以SARS
coronavirus Hong Kong/03/2003 RNA-directed RNA polymerase gene做為例子---
利用電腦來模擬用restriction
enzyme作用在peptide下的結果。例如:以SARS
coronavirus Hong Kong/03/2003 RNA-directed RNA polymerase gene做為例子---
![]()
![]()




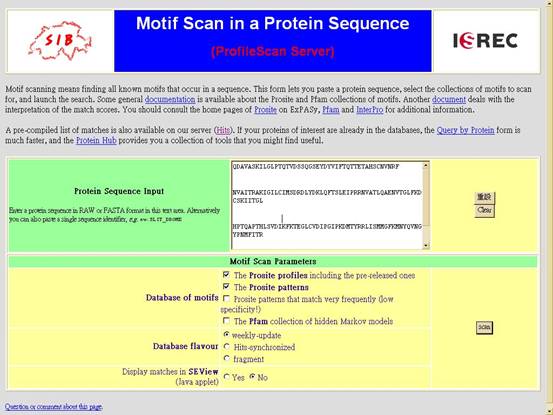
![]()

做MotifScan,從輸入的序列和在database中已知的motif做比對
![]()
![]()


![]()

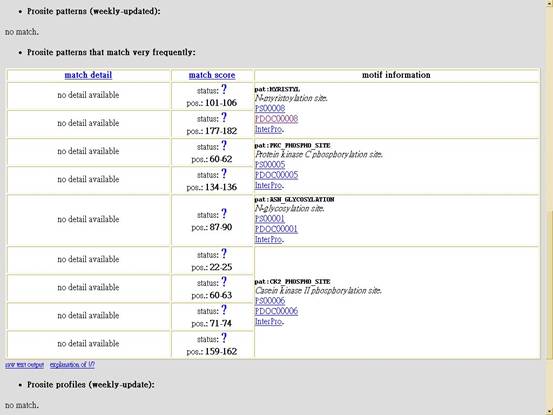
![]()

用前面所使用的SARS序列來做例子

在只有勾選prosite profiles和prosite pattern兩個時,出現的結果是no match。
在加上勾選Pfam及Prosite patterns that match very frequently (low specificity!)後,得到和ScanProsite及Prosite scan相同的結果。
![]()
![]()





若是在MotifScan頁面中勾選「Display matches in SEView」,則會出現:
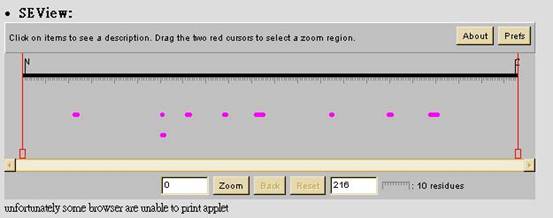
它利用Java applet的技術,來表示輸入序列中的motif部分。
用“NNPredict”來測試蛋白質的二級結構
![]()
![]()

![]()

![]()
![]()
![]()

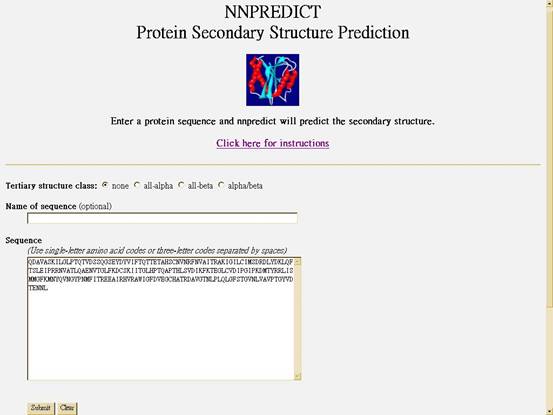
結果一:在Tertiary structure class的選項,選擇「none」

圖中的H是代表helix;E是代表strand;-則是沒有預測。預測出來的值是指在這段序列中,有哪些amino acid會是出現在helix,哪些會是出現在strand。
結果二:在Tertiary structure class的選項,選擇「all-alpha」

可以明顯看到它就只有顯示出helix的部分
結果三:在Tertiary structure class的選項,選擇「all-beta」

和結果二不同的是,它只顯示出strand的部分
結果四:在Tertiary structure class的選項中,選擇「alpha/beta」

下面再用另一種工具來做蛋白質二級結構的預測--------Jpred
![]()

![]()

![]()


![]()
![]()

![]()


![]()
Raw protein sequence
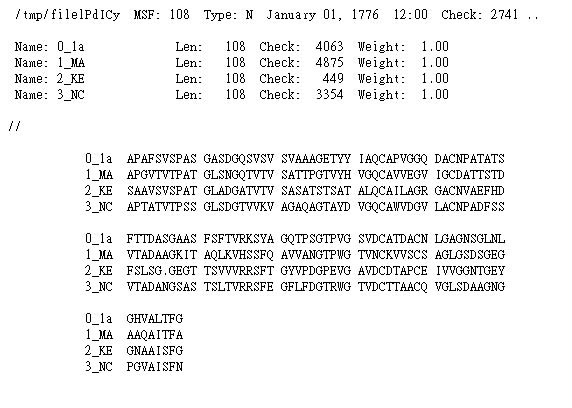
MSF format

現在以先前用的序列做為範例來測試


它會出現這些選項,可以選擇想以怎樣的型式來觀看所得到的結果。
第一個選項是以瀏灠器外掛Jalview來觀看;第二個則是把結果下載到電腦,用電腦中安裝的Jalview觀看;第三個是直接用Html型式看所得到的結果、…,其中,第七個選項是Unix作業系統所使用的壓縮格式。在這裡採第一個來顯示,直接按視窗中的「JalView」按鈕,電腦會另外出現一個Jalview的視窗。

在這視窗內,有多種的應用選項可以使用,例如把「Helix propensity」用顏色表現出來。
![]()

依照想看出怎樣的結果,來設定它所顯現的內容。
此外,若是用Html的選項來看結果,則會得到下面的網頁:

它和Jalview不同的是,沒有多種的屬性可以應用在顯示出不同的特性值。
在網頁的下半部有指出上半部的一些名詞所分別代表的意思。
PSIpred------蛋白質二級結構的預測
![]()
![]()

![]()
![]()



![]()
![]()


![]()



這是以先前SARS序列所做的結果,點選最後的連結可以進到下面的畫面。

可以點選PostScript File;PDF File;JPEG Page 1,用這些來看所得到測試的結果。在這裡以JPEG為例。

其中綠色圓柱狀是表示helix,黃色箭頭是表示strand,黑色直線是coil的部分。以這樣的模式來顯現出測試模擬的結構。