蛋白質結構與功能
整理: 蘇虔瑩、邱子玲、尤明村、蘇宿睿
7.在眾多可能的Polypeptide Chains中只有少數對生物有用
9.能被蛋白質採納的蛋白質摺疊(Protein Folds)有限
11.透過計算的方法(Computational Methods)能將胺基酸序列與已知的Protein Folds做配對
12.某些Protein Domain稱為模組(Module),可形成許多不同蛋白質的某個部分
14.大的蛋白質分子通常包含一條以上的polypeptide chain
15.有一些蛋白質以長螺旋絲(Helical Filament)方式存在
18.蛋白質分子通常擔任結合成大分子結構的次單位(Subunit)
20.複合生物結構(Complex Biological Structures)的形成通常需Assembly Factors的協助
1.前言
經由觀察蛋白質可了解一個細胞的電位與生化活性。細胞中蛋白質占絕大多數,除具有阻隔的功能外,更執行了細胞內所有有關酵素的作用,並推動許多化學反應的進行。埋藏在細胞膜上的蛋白質,會形成通道或是幚浦,以控制小分子進出細胞。另外也有些蛋白質,在細胞間擔任傳遞訊息的角色,或是整合訊號將之由細胞膜傳至細胞核。還有一些小分子蛋白如:topoisomerase,能解開糾結的DNA;其他特化的蛋白,包括antibodies、toxins、hormones、antifreeze molecules、elastic fiber、ropes及luminescence的來源。在我們希望了解基因如何運作、肌肉如何收縮、神經電位的傳導、胎兒發育及身體各種功能之前,必須先進一步了解蛋白質。
以下我們將探討各種不同胺基酸所在的位置,如何影響一個蛋白質形成特定的三度空間結構,並更進一步地了解此特定結構與其功能之間的相互關係。
蛋白質包含有20種不同化學特性的胺基酸,一分子的蛋白質是由一長鏈胺基酸所組成,藉由共價胜肽鍵相互連接,因此也稱之為polypeptides。
每種蛋白質都有其獨一無二的胺基酸序列。沿著核心由重複序列所形成的polypeptide chain稱為polypeptide backbone;而附於此骨架上的周邊結構(即胺基酸的side chain),則給予每個胺基酸不同的特性(圖1)。這些side chain部分不具極性,呈疏水性;部分則帶有電荷,可能為正電或負電。20種胺基酸的原子結構,請見圖2;而縮寫表示法,請見圖3。

圖1 : 蛋白質的結構組成。一蛋白質包含了一條polypeptide backbone,與其上附著的side chains。每一種蛋白質的胺基酸序列及數目都不同,也就是這些具有不同化學特性的side chain造成了每種蛋白質間的差異。polypeptide chain的兩端分別帶有游離的胺基(amino group)及羧基(carboxyl group),各稱為amino terminus(或N-terminus)和carboxyl terminus(或C-terminus)。

圖2
: 20種胺基酸的原子結構。
圖2(續): 20種胺基酸的原子結構。

圖3 : 20種胺基酸的縮寫表示法以及side chain 的特性。
原子就像是堅硬的球體,且有一定的凡得瓦半徑。因為原子間不能重疊的關係,大大地限制了多肽鏈中可能的bond angles(圖4)。此限制加上其他空間上的交互作用,則嚴格的限制了原子在三度空間中的排列變化。然而,一條長的flexible chain,例如蛋白質,仍然能以非常多的方式進行摺疊。另外,蛋白質鏈的摺疊也受到一些非共價性鍵結的限制;非共價鍵結有3種,分別是氫鍵、離子鍵與凡得瓦作用力。共價鍵的強度約是非共價鍵強度的30至300倍,但很多的非共價性鍵結一起作用,卻能使兩個polypeptide chain緊密的結合。因此,摺疊結構的穩定性是由大量非共價性鍵結所組成的強度來決定。
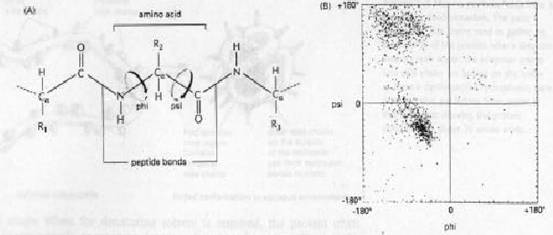
圖4
:
多胜肽鏈中,空間限制對bond
angles的影響。
(A)
每一個胺基酸貢獻三個鍵給polypeptide
backbone,而胜肽鍵(peptide
bond)為平面的一部分,無法轉動。然而,Cα-C
鍵和N-Cα
鍵則可以轉動,轉動的角度分別稱為psi
(Ψ)和phi
(ψ)。R
group表示side
chain的部分。
(B)
蛋白質主鏈原子的構形決定於每個胺基酸的ψ、Ψ
angle。因為胺基酸內的原子之間會有空間上的障礙,因此大多數的ψ、Ψ
angle
pair並不會發生。在Ramachandran
plot中,每一個點表示在蛋白質中觀察到的一對ψ、Ψ
angle。
另外還有一種微弱的作用力,也對蛋白質的構形有重要的影響。如同先前所提到的疏水性分子,包括部分非極性胺基酸,於水的環境下會聚集在一起,以減低其受水分子氫鍵分散力的作用。因此,胺基酸的極性與非極性,亦是決定蛋白質摺疊構形的重要因素。非極性胺基酸包括有phenylalanine、leucine、valine及tryptophan,它們傾向於聚集在分子的內部 (如同油滴於水中一樣),這樣能避免其存在於細胞內與水的接觸。而極性胺基酸則有arginine、glutamine和histidine,它們會傾向於排列在分子的外側,而能與水或是其他極性分子形成氫鍵。(圖5)當蛋白質內部有極性胺基酸存在時,通常會與其他極性胺基酸或是polypeptide backbone形成氫鍵。(圖6)
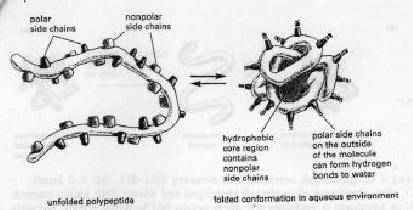
圖5
:
蛋白質如何摺疊成一緊密的構形。具有極性side
chain的胺基酸傾向聚集在蛋白質的外側;而具有非極性side
chain的胺基酸則埋藏在蛋白質內部,形成一緊密包裹的疏水性核心(hydrophobic
core)。圖中的蛋白質約含30個胺基酸。
圖6
:
蛋白質分子中的氫鍵。在已摺疊之多胜肽鏈(folded
polypeptide chain)內的鄰近區域間會形成很多的氫鍵,以幫忙穩定三度空間的構形。圖中顯示的蛋白質為酵素lysozyme的一部分。
由於胺基酸不同的排列所產生之交互作用力,使得每種蛋白質具有其特殊的三級結構,最終摺疊所形成之構形擁有最小的自由能(free energy)。研究蛋白質的摺疊,需利用高度純化的蛋白質。藉由某些溶劑的處理,能將folded chain之間的非共價性鍵結打斷,使蛋白質被展開(unfolded)或變性(denatured),而喪失其原有的形狀,變成一flexible的多胜肽鏈(polypeptide chian)。當溶劑移除後,蛋白質通常會自動地摺疊(refold)回原來的形狀,此過程稱做renature(圖7)。此表明了:主導蛋白質三維構形的所有資訊,皆包含在其胺基酸的序列裡。
圖7
:
變性(denatured)蛋白質的重新摺疊(refolding)。
(A)
此實驗證明了一個蛋白質的構形只決定於其胺基酸序列。
(B)
尿素的結構。尿素在水中的溶解性很高,而且濃度高的時候可以將蛋白質解開(unfold)。
一般情況下,蛋白質會摺疊成單一穩定的構形,此構形會在與其他分子作用時產生輕微的變化,而這與其功能有關。雖然蛋白質在無任何外在的幫忙下,即能摺疊成其正確的構形;但在活體細胞內,蛋白質的摺疊通常會受到molecular chaperones的幫忙。此類蛋白會結合至已部分摺疊的多胜肽鏈,並幫助其形成最適當的摺疊構形。存在於細胞質的chaperones非常重要,它能防止新合成的蛋白質暴露出疏水性區域而相互作用形成沉澱。然而,蛋白質最終的三度空間結構,仍是由胺基酸序列所決定,chaperones只是使摺疊的作用進行的更確實。一個大的蛋白質分子通常由許多不同的domains構成,這些結構單位(domains)的摺疊部分是相互獨立的。而蛋白質結構相當複雜,因此要簡化地描述一個蛋白質結構,可以透過很多不同種的方式,而每一種描述方式,都是用來強調此蛋白質不同的特徵。
早於五十年前研究頭髮和蠶絲時,就發現所有的蛋白質三級結構中,一般包含有α helix和β sheet。最早發現的摺疊模式是在α-keratin 中發現的α helix,其他如皮膚及其衍生物,像是頭髮、指甲和角的蛋白質,多含此種摺疊模式。相隔一年後,於fibroin(silk的主成分)中發現第二種摺疊模式,稱為β sheet。這兩種摺疊模式會這麼普遍,是因為polypeptide backbone中的N-H和C=O間的氫鍵所造成的,跟side chain的交互作用無關,所以導致這兩種結構可以出現在許多不同的序列之中。如圖8A及8D。
蛋白質的核心部分多為β sheet,兩相鄰的polypeptide chain可以同方向或者反方向的方式,藉由氫鍵的力量形成堅固的結構,如圖9及8D。α helix 為單一polypeptide chain旋轉成堅硬的柱狀體,而每隔四個peptide bond就可於N-H和C=O間形成氫鍵,所以每隔3.6個胺基酸,可旋轉上升形成一固定的旋轉體,如圖8A。
細胞膜中的transport protein和receptor中,α helix的結構相當多,通常藉由短的α helix摺疊模式貫穿細胞膜的雙層結構。而α helix是由大量的非極性胺基酸所組成,其非極性的side chains會露在外頭,而極性的polypeptide backbone則會躲在裡頭。
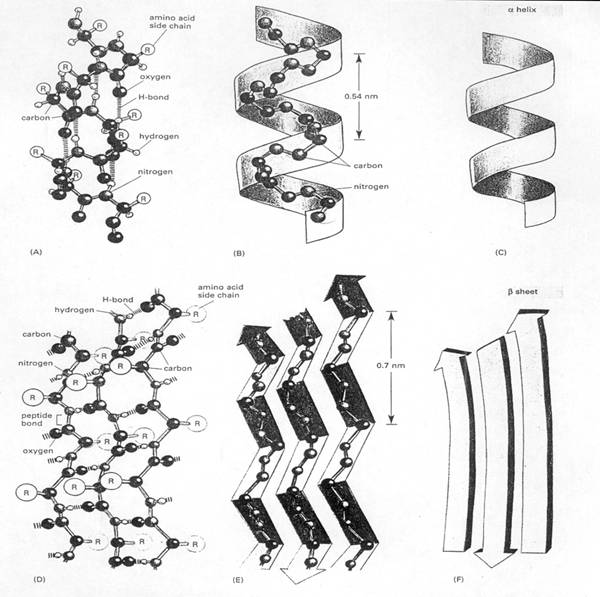
圖8
: α helix和β
sheet中polypeptide
backbone的規則構形。
(A、B、C)
為α
helix,(D、E、F)
為β
sheet。此例中的β
sheet為antiparallel。
(A、D)
顯示了polypeptide
backbone的所有原子,但side
chain的部分以R表示之。(B、E)
只顯示backbone上的原子。(C、F)
則是α
helix和β
sheet的簡略表達方式。
另有其他蛋白質,其α helix和α helix會相互纏繞形成一特殊的穩定結構,稱為coiled-coil。會形成這種結構是因為此兩個α helix含多數非極性(厭水性)的胺基酸,所以兩兩相互纏繞,使非極性的side chain包覆於兩α helix間的內側,形成較為長形的蛋白質,如α-keratin,可提供皮膚的強韌度;而肌凝蛋白亦為如此,以提供肌肉收縮的能力,如圖10。
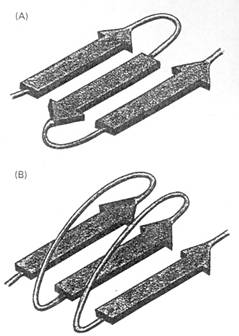
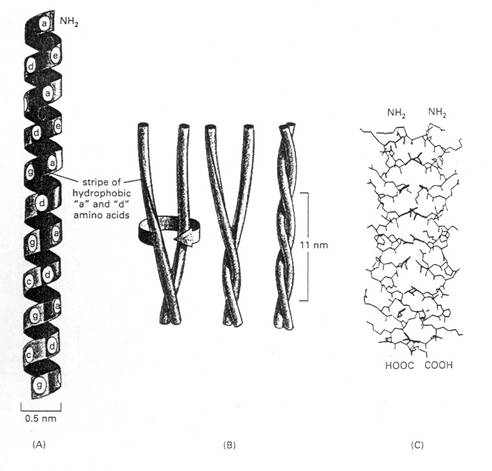
圖9
: β sheet的兩種形式。
(A) antiparallel β sheet。
(B) parallel β sheet。此兩種形式在蛋白質中都很常見。
圖10 : coiled-coil的結構。 (A) 由 “abcdefg”七個胺基酸相繼排列所形成的αhelix。胺基酸 “a”和 “d”在圓柱狀的表面靠的很近,彼此可連成一條稍微彎曲的線。會形成coiled-coil結構的蛋白質都具有非極性的胺基酸就像 “a”和 “d”的位置一樣。 結果就像 (B)圖所示,兩條α helix會藉由非極性的side chain的交互作用而互相纏繞;而極性的胺基酸則被暴露在水溶液的環境中。 (C) 利用x-ray crystallography所測出的coiled-coil之原子結構圖。
蛋白質是由無數的原子所構成,而原子間則藉由共價鍵和非共價鍵來連結,而形成一複雜的三度空間結構。目前有電腦軟體可提供三度立體空間的圖像,以呈現各種不同旋轉角度的立體結構。生物學家把蛋白質結構分成四個等級:一級結構(primary structure)指胺基酸的序列;二級結構(secondary structure) 由α helix和β sheet所構成;三級結構(tertiary structure)指的是整個polypeptide chain在三度空間形成的樣子;而如果一個蛋白質是由兩個以上的polypeptide chain所組成,則最終形成的結構稱為四級結構(quaternary structure),如圖11。Protein domain為一部分的polypeptide chain所形成之獨立、緊密、穩定的小結構,通常約為40至350個胺基酸,在大分子的蛋白質中可視為一基礎單位,而不同的domain提供不同的功能。

圖11
:
由四個domains形成的蛋白質(Src
protein)。其中兩個domains形成一protein
kinase enzyme,而SH2和SH3
domain則執行調節的功能。
(A)
以ribbon
model表示之,並包含一ATP受質(substrate)。
(B) spacing-filling model。Src
protein與ATP結合的位置是在兩個kinase
domain的交界處。SH2
domain的詳細結構可參見圖16。
最小的蛋白質只含一個domain,而大分子的蛋白質含數十個domain。一個domain可能由數個α helix、β sheet或兩種摺疊形式的組合所構成,這些不同的組合都稱為protein fold。截直目前為止,發現在10,000個已知詳細結構的蛋白質中,約含有1000個不同的protein fold。
7.在眾多可能的Polypeptide Chains中只有少數對生物有用
胺基酸共有20種,故4個胺基酸就有20x 20x 20x 20x =160,000種可能的組合方式。理論上,應該有相當多種不同的polypeptide chain 產生,但只有相當少的形式可以有穩定的三度空間結構,其機率約為百萬分之一。事實上,在細胞中的所有蛋白質都會採取獨特且穩定的構形存在。原因是天擇(natural selection)的作用,使對細胞存活沒有幫助的蛋白質被淘汰掉。天擇的作用不只使現今的蛋白質擁有如此的胺基酸序列所以可以有一非常穩定的構形;而且也使此構形有其化學特性,得以在細胞中執行特定的催化或結構上的功能。蛋白質的構築十分精準,即使是一個胺基酸改變了幾個原子,有時候也會破壞整個分子的結構,而造成功能的喪失。
所謂protein family,是指一群胺基酸序列和三級結構都非常相似的蛋白質。Serine proteases 為一個蛋白質水解酵素的大家族,包含消化酵素chymotrypsin、trypsin、elastase和一些與血液凝結有關的protease。雖然這些蛋白質的胺基酸序列和三級結構都非常相似,如圖12,但其酵素活性、分解的蛋白質或peptide bond皆不同。其他蛋白質家族亦是如此,但也有許多蛋白質家族,其成員的胺基酸序列差異性較大,因此不能只依據胺基酸序列來歸納其所屬的蛋白質家族,必須配合其三度空間的結構,才可歸納分類之。例如:酵母菌的α2 protein和果蠅的engrailed protein,此兩者都是基因調節蛋白(gene regulatory protein),屬於homeodomain family的成員。但是,在它們60個胺基酸長的序列中只有17個胺基酸相同,所以只有在比較其三度空間結構的時候,才能得知兩者的相似性,如圖13。
同一家族的蛋白質成員,常常有不同的功能。原因是演化過程中的篩選作用,讓有部分胺基酸序列改變的成員,在生物活性上產生有用的變化,使得每個家族成員在功能上都有各自不同的特性。但也有許多胺基酸的改變,並不會造成蛋白質結構或功能上的影響。
當一生物的基因體序列(genome sequence)被定出來後,蛋白質家族便能很容易地被辨認。例如:經由DNA序列的測定,發現在線蟲綱Caenorhabditis elegans九千七百萬鹼基對的genome中,包含有超過18,000個基因。再透過序列的比對,發現這些基因的產物,包含許多屬於不同protein family的domains,例如:有388個基因含有protein kinase domains、66個基因含有DNA和RNA helicase domains、43個基因含有SH2 domains、70個基因含有immunoglobulin domains,以及88個基因含有DNA –binding homeodomains。
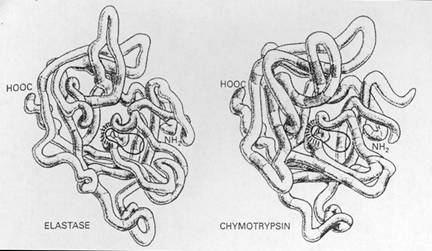
圖12 : 比較兩種serine protease的構形。圖上所顯示的為elastase和chymotrypsin的脊骨構形(backbone conformation)。這兩個酵素的構形非常相似。圈起來的部分表活性區域(active site),為與受質結合的地方,並利用水解的方式將受質的胜肽鍵切斷。
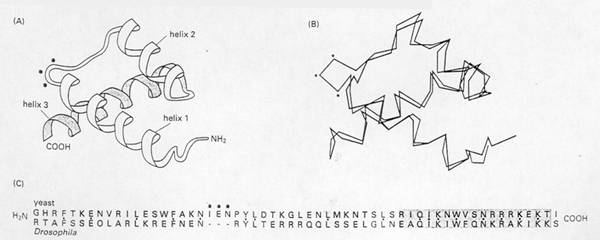
圖13
:
兩個同屬Homeodomain的蛋白質之比較。
(A) ribbon model。此兩蛋白質共通的結構。
(B)
α-carbon的位置軌跡。藉由x-ray
crystallography的方式測定酵母菌的α2
protein和果蠅的engrailed
protein之三度空間結構。
(C)
胺基酸序列的比較,黑點表完全相同的位置。
9.能被蛋白質採納的蛋白質摺疊(Protein Folds)有限
有關於細胞的新知進展的很快,在1950年代的時候,對蛋白質的胺基酸排列還不是很清楚。到了1960年代,第一個蛋白質三度空間結構,經由X-ray結晶繞射法被測定出來。至於現在,要將基因序列轉成蛋白質序列,並產生大量純的蛋白質已經不再是大問題了,這都要歸功於DNA轉殖技術及基因工程技術所賜。但是,蛋白質三度空間結構的決定上,仍存在著許多的困難。因為利用X-ray繞射的技術時,蛋白質必須先養成晶體(protein crystal),而並不是所有的蛋白質都能養成晶體,所以只能靠著trial-and error的方法去試,直到成功為止。
膜蛋白以及大蛋白複合物,因為具有可移動的部分,所以很難養成晶體,因此這類蛋白質的結構知道不多。後來,將一個大蛋白質的結構先分成許多個別的domains後,再加以分析,並藉由X-ray結晶繞射法與核磁共振儀(NMR)的結合,現在已能解出更多不同蛋白質的三度立體空間結構。
目前的資料庫中,已包含了五十萬筆以上的已知蛋白質序列,而且隨著越來越多基因體(genomes)被定序出來,資料還在以非常快的速度增加中。強有力的電腦搜尋程式,能將每個新發現的蛋白質,與資料庫中的所有資料做比對,尋找可能的相關性。同源蛋白質(homologous proteins)是指一群蛋白質其基因來自同一個共同的ancestral gene,而這些蛋白質的胺基酸序列,在統計上必須有顯著的相似性才行。
因為資料庫裡的蛋白質序列非常多,搜尋程式會找到許多nonsignificant match的資料,造成一背景值的雜訊,以致於很難挑出所有的資料,只能挑出相關性最高的部分。一般來說,兩個蛋白質的序列,相似度須達30%以上,才能確定會成為符合的資料,而被挑選出來。然而,許多短的signature sequences(”fingerprints”)為特定蛋白質功能的指標,這些指標序列能廣泛地用於找到同源性較遠的序列(圖14)。
這些蛋白質的比對非常重要,因為相關的結構常常意味著相關的功能。因此,如果發現一新的蛋白質其胺基酸序列,與一已知功能的蛋白質有同源性,則便能省下許多年的實驗時間。
圖14 : 可利用短的signature sequence以尋找同源的protein domains。兩個短的胺基酸序列,分別為15和9個胺基酸長,就能利用來在廣大的資料庫中搜尋出SH2 domain。此處列出來的是人類與果蠅的Src protein之SH2 domain的前50個胺基酸。比較的結果,序列相同的位置以一個字母的胺基酸縮寫表示,若是相近的就以+號表示,而完全不同的則以空白表示。
11.透過計算的方法(Computational Methods)能將胺基酸序列與已知的Protein Folds做配對
我們知道可以透過很多的方式使蛋白質具有相同的三度空間結構;而且在演化的過程中,會發生許多隨機的突變,使得胺基酸的序列產生改變,但並沒有造成蛋白質構形(conformation)上的重大改變。因此,結構生物學家想要測定出蛋白質在自然狀況下,可能含有的所有protein folds,並且設計出一套以電腦為基礎的方法,能將某一胺基酸序列與先前已測定出構形的domains做比對,以檢測出此一胺基酸序列可能代表的domain為何。
計算的方法稱為 “threading”,能將一胺基酸序列與特定的protein fold做配對。電腦會從每種可能的folds中,搜尋出與此胺基酸序列最合適的結構,並以數值化的分數來反映其結構穩定性的評估結果。
某些胺基酸序列能找到很適合的三度空間結構;然而,有些胺基酸序列,在已知的protein folds中,並不能找到可能相符的一種。所以結構生物學家必須利用x-ray與NMR的方法對這類蛋白質做研究,以期能找出更多的protein folds,而能建立一個含有完整library的資料庫,包含有自然界中存在的所有protein folds。有了這樣的library後,再加上計算方法的改進,可能就能使一蛋白質在知道其胺基酸的序列後,很快地就能得到其最接近的三度空間結構。
12.某些Protein Domain稱為模組(Module),可形成許多不同蛋白質的某個部分
如同先前的陳述,大部分的蛋白質是由一系列的protein domains所構成,這些domains是由polypeptide chain的不同區域各自摺疊而形成的緊密結構。此類multidomain proteins的產生,應是由於encode各個domain的DNA序列意外地相接在一起,而創造了新的基因。新的結合表面(binding surfaces)常常是因為domains互相並列而產生。許多大的蛋白質被發現是由已存在的domains以新的組合方式而產生的,此過程稱為domain shuffling(圖15)。有一些protein domains在演化過程中特別容易移動(mobile),這些domains稱為protein modules,通常會小於一般平均的domains,約40-200個胺基酸長;而且它們似乎擁有特別容易變化的結構。SH2 domain即為一例(圖16),而圖17中也顯示了幾個protein modules的結構。
每個module都有由β strands形成的穩定核心結構,而較不整齊的loops則從此處突出。這些loops會形成與其他分子結合用的binding sites,例如immunoglobulin fold,為構成抗體分子的基本部分。這類以β sheet為基礎的modules能成功地演化,是因其提供了很方便的構造,只要對突出的loops做小小的改變,就能產生新的binding sites。
Protein module的另一個特徵是可以很容易地插入其他的蛋白質中。圖17中有五個modules的N端和C端位在相對的兩極。此種 “in-line”的排列方式,意味著當DNA encode這類module時發生了tandem duplication,而duplicated modules則可以很容易地連結成串以形成extended structure(圖18)。Stiff extended structure是由一系列模組所組成,尤其在細胞外基質分子及細胞表面的接受器之extracellular portion最常見。其他模組包括SH2 domain及kringle (圖17)。
圖15 : domain shuffling。蛋白質在演化的過程中會發生protein domain的shuffling。Chymotrypsin由兩個domain所形成。而下面的三種protease則具有高度的調節性,而且也較具專一性,它們的protease domain會與其他的domains相連接,可能是一個或一個以上與EGF同源的domain,或calcium-binding protein domain或 “kringle” domain。
圖16 : SH2 domain的四種描繪方式。
圖17 : 某些protein modules的三度空間結構。球形的部分表N端和C端。
圖18 : 由鏈續的protein module所形成的延伸結構(extended structure)。圖為fibronectin中的四個fibronectin type 3 modules。(A) ribbon model。 (B) spacing-filling model。
人類的染色體只包含了30,000-35,000個基因。就基因數目而言,其複雜度不到線蟲的2倍之多。而基因組(genome)的序列也顯示了,脊椎動物的protein domains幾乎全來自無脊椎動物,人類的domains中只有7%是屬於脊椎動物所特有的。然而,平均來講我們人類的蛋白質複雜度較高。在脊椎動物演化的過程中發生了domain shuffling,引起許多protein domains產生新的組合,結果造成人類蛋白質中protein domains的組合幾乎是在蟲或蒼蠅中發現的兩倍。舉例來說,在人類蛋白質中的trypsinlike serine protease domain至少與18種其他的protein domains發生連結,然而在蟲體中只發現有5種不同domain會以共價鍵鍵結方式與此domain結合。這項額外的變化,大大地增加了人類的蛋白質之間交互作用的範圍。
14.大的蛋白質分子通常包含一條以上的polypeptide chain
protein chain靠著微弱的非共價鍵以摺疊成特定的構形(conformation),而蛋白質之間也同樣地藉由這些非共價鍵來相互連結,在細胞內形成較大的結構。蛋白質表面上的任一區域若能與另一分子藉由非共價鍵產生作用,則此區域稱為結合位(binding site)。假使一個蛋白質的binding site能辨識另一蛋白質的表面,則兩個folded polypeptide chains之間會在幾何位置上產生緊密的結合,而創造出一個更大的蛋白質分子。這類大型蛋白質中的每一條polypeptide chain,都稱做蛋白質次單位(protein subunit)。舉一簡單例子說明之:Cro repressor protein為細菌中的一種基因調節蛋白,可結合到DNA上,而關掉基因的作用。而此蛋白質是由兩個一模一樣的folded polypeptide chain以頭對頭的方式結合在一起,形成的對稱性複合物。這兩個相同的folded polypeptide chain 即為protein subunits。此外,neuraminidase是由四個相同的protein subunits所組成,彼此以頭對尾的方式排列,而形成一環形結構(圖19)。
細胞中很多蛋白質包含有兩種或兩種以上的polypeptide chain。如血紅素(hemoglobin),它存在於紅血球中攜帶氧氣,是由兩條相同的α-globin subunits和兩條相同的β-globin subunits 所組成(圖20)。這類由multisubunit 所組成的蛋白質,在細胞中相當常見,而且它們可以非常的巨大。
圖19 : 由多個相同protein subunit所組成的蛋白質分子。酵素neuraminidase的環狀結構,是由四個相同的多肽鏈所組成的。
圖20 : 由兩個不同的subunits以對稱方式組合成的蛋白質。血紅素是紅血球中很豐富的蛋白質,包含了兩個α globin和兩個βglobin。而這四個多肽鏈分別都含一個heme分子,此為氧所結合的位置。因此,血液中每一個血紅素分子可以結合四個氧。
15.有一些蛋白質以長螺旋絲(Helical Filament)方式存在
有些蛋白質分子能夠組合起來形成絲狀的結構,其長度可以很長,甚至橫跨整個細胞。如果一個分子上具有一個binding site可以與同一分子上的另一區域的表面做結合,則這些相同的分子便能相互串聯成一條很長的chain(圖21)。例如actin filament,由許多actin蛋白分子,組成長的螺旋狀結構(圖22)。為什麼螺旋狀結構(helix)在生物學中這麼地普遍?就如我們知道的,生物性結構常常是把許多非常相似的subunits連接起來,而形成一長的重複性鏈狀結構(repetitive chain)。如果所有subunits都一樣,則相鄰的subunit間只能以單一種方式做連接,以使自由能降到最低。結果每一個subunit就這樣依序地一個接一個,以同樣的方式連結在一起;然而這些subunits很少會連接成一條直直的chain,而是形成螺旋狀的結構(圖23)。依照螺旋階梯的旋轉方式,可分為右手旋(right-handed)和左手旋(left-handed)。Helix在生物結構中非常常見,subunits間或是由小分子藉由共價鍵相互連接(例如:amino acids in an α helix),或是由較大的蛋白質分子藉由非共價鍵相互連接(例如:actin分子連接成actin filament)。
圖21 : 蛋白質的組合(protein assemblies)。 (A) 只具有一個結合位置(binding site)的蛋白質,可以和另一個相同的蛋白質形成dimer。 (B) 一蛋白質上若具有兩個不同結合位置,則許多相同的這些蛋白質分子通常會形成一條長的helical filament。 (C) 如果兩個binding site的位置彼此很適合,則可能會形成一封閉的環形結構。
圖22 : Actin filament。 (A) actin filament的穿透式電顯圖,以負染色的方式染色。 (B) actin filament上的actin 分子是以螺旋的方式排列。
圖23 : Helix結構的某些特性。 (A-D) 當一連串的subunits以規律的方式相互結合後,則會形成一螺旋狀的結構。(A)、(B)、(C、D)的α helix,每一圈的subunit數分別是2個、3個和6個。(E) helix可以有兩種旋轉方式,一為右手旋(right-handed),一為左手旋(left-handed)。
到目前為止,我們討論的蛋白質皆為球狀蛋白,而球狀蛋白是由多胜肽鏈(polypeptide chain)疊合成緊密的球狀,且擁有不規則的表面。酵素多為球狀蛋白:即使許多是由許多subunits組成,不僅巨大且複雜,不過多數整體而言呈圓形。相反的,其他蛋白質因細胞需要各個蛋白質分子伸展很長的距離,通常擁有較為簡單且細長的三度空間結構,我們一般稱之為纖維狀蛋白(fibrous proteins)。
細胞內纖維狀蛋白的其中一個大家族便是由α-keratin及其親屬(relatives)所組成。角蛋白纖維(keratin filaments)非常的穩定,且是存活時間很長的結構,如頭髮、角及指甲之主要成分。α-keratin分子是由兩個相同的subunit組成的二聚物(dimer),每個subunit各為一長的α helix,兩者互相纏繞而形成coiled-coil。在這些coiled-coil區域的兩端,便由包含結合位置的globular domains所覆蓋。這使得這類的蛋白質能組合成繩索狀的intermediate filaments,此為細胞骨架(cytoskeleton)的重要組成。
纖維狀蛋白在細胞外特別豐富,它們是膠狀的胞外基質(extracellular matrix)的主要成份,用以幫助聚集在一起的細胞相互結合而形成組織。胞外基質蛋白經由細胞分泌到外圍,而在那裡通常會組合成薄板(sheet)或長纖維。膠原蛋白(collagen)在動物組織中是這類蛋白含量最多的。一個膠原分子由三個長的多肽鏈構成,每一多肽鏈的第三個位置皆為非極性的胺基酸-glycine。這個規則的結構,讓鏈與鏈之間能互相纏繞,而形成長的規則三螺旋(triple helix)(圖24)。接著,許多的膠原蛋白分子彼此以側面或頭尾方式相互結合,而形成非常堅韌的collagen fibrils,以提供結締組織的張力。
胞外基質中和膠原蛋白全然相反的一種蛋白質為彈性蛋白(elastin)。彈性蛋白分子是由相當鬆散且無結構性的多肽鏈,經由共價鍵結合成橡膠般具有彈性的網狀物。和多數蛋白不同的是,它們不含有獨特的穩定結構,但能夠從一個構形回復到另一個構形(圖24B)。這使得彈性纖維能讓表皮及其他組織,如動脈與肺臟,能伸展與反彈而不會有撕裂發生。

圖24 : 膠原蛋白(collagen)與彈性蛋白(elastin)。 (A) 膠原蛋白呈現triple helix的結構。許多桿狀的collagen分子相互的交錯連結而在細胞外的空間(extracellular space)形成collagen fibrils。(B) Elastin polypeptide chains相互交錯連結而形成橡膠似的彈性纖維(elastic fibers)。當elastin fiber拉直時,每個elastin分子也會伸展開來,一旦展開的力量鬆弛,則每個分子便會自動地捲回去。
許多蛋白質分子不是附著於細胞膜外,便是被分泌出去成為胞外基質的一部分。所有的這種蛋白都直接的暴露在胞外的狀態下。為了要維持它們的結構,這類蛋白的多肽鏈常藉由共價鍵交錯連結而達到穩定。這些連結能將兩個胺基酸綁入一個蛋白質內,或是將兩個不同的多肽鏈結合在一個多單元(multisubunit)的蛋白質內。在蛋白內最一般的交錯連結便是雙硫鍵的共價鍵鍵結。雙硫鍵形成於蛋白質即將準備輸出到膜外時,它們主要是在內質網內藉由一個酵素催化而形成,此酵素能將已摺疊的蛋白質內兩個鄰近的cysteine支鏈上的-SH group連結在一起(圖25)。雙硫鍵並不會改變蛋白質的構造,但卻會扮演類似atomic staples的作用,去強化其最偏愛的結構。舉例來說,lysozyme(一種在淚液中能分解細菌細胞膜的酵素)就是因為藉著這樣的交錯鍵結而達穩定,才能長期保有它的抗菌活性。
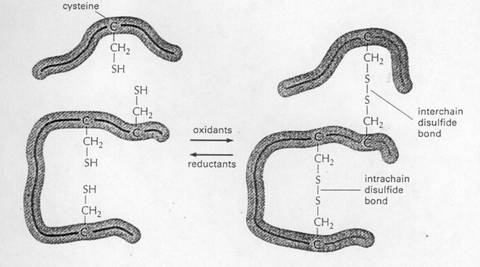
一般而言,雙硫鍵在細胞質內無法形成,因為在細胞質內有高濃度的還原劑,會將雙硫鍵轉變回含有-SH基團的cysteine。顯然地,在胞內這樣溫和的環境下,蛋白質並不需要這種形態的強化。
圖25 : 圖中所顯示的為相鄰的Cysteine side chains如何形成共價的雙硫鍵結。這些鍵結可以連結一條polypeptide chain裡的不同部分,也可以連結兩個不同的polypeptide chains。因為打斷一個雙硫鍵所需的能量比打斷整套的非共價鍵所需的能量還大的多,所以雙硫鍵可以提供蛋白質重要的穩定力量。
18.蛋白質分子通常擔任結合成大分子結構的次單位(Subunit)
一個蛋白質分子能和自我結合形成環或纖維在細胞內產生更大的結構-超分子結構,如酵素複合物、核醣體、蛋白纖維(protein filaments)、病毒、胞膜等。這些大物體並非由單一、巨大、共價鍵接合的分子所形成,其實它們是由許多獨立製造的subunit藉由非共價鍵結合而成。
利用小的subunit來建築大結構有幾個優點:
1. 由單一或一些重複的小subunit來構成一個大的結構僅需小量的基因資訊。
2. 因為subunits間是由許多能量較低的鍵結相互組合,所以組構(assembly)或解構(disassembly)可被迅速的控制及回復。
3. 結構組合中發生的錯誤更容易被避免,因為在組合的過程中,修正機制可以作用而排除畸形的subunits。
有些蛋白質次單元(protein subunits)利用六邊形圖樣組合成平板。特化的膜蛋白有時候在脂質雙層膜(lipid bilayers)中會有如此的排列。而各個subunit在幾何形態上只要有些微的改變,六邊形的平板便能轉變為管狀(圖26),或者,如果改變的更多,便會形成一個中空球體。能與特定的RNA及DNA分子結合的蛋白質管和球便成為病毒的外套。
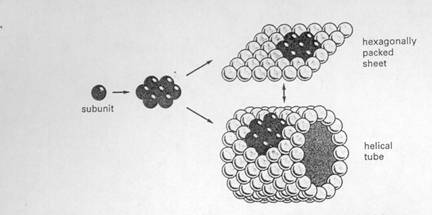
圖26 : 一個由單個protein subunit開始組合(assembly)的例子。此過程需很多蛋白質之間的接觸。球狀蛋白聚成的六角形單元,能形成平板或管子。
封閉型的結構,如環、管、球體,能提供額外的穩定性,因為它增加了兩個protein subunits間的鍵結數。此外,因為這樣的結構是由subunit間的交互作用而成,因此,只要對各個subunit有些小改變便能趨使整個大結構的組構(assemble)亦或是解構(disassemble)。這樣的原則在許多簡單的病毒的中空球狀蛋白質外套(protein coat)或capsid上最能生動的闡述。capsid通常由數以百計的相同protein subunits所構成,用以包圍和保護病毒的核酸(圖27)。capsid的蛋白質必須擁有一個能適時改變的結構:它必須有數種不同的接觸面以構造球體,且還能改變排列讓核酸跑出,以便一旦病毒進入寄主細胞就能起始病毒的複製。
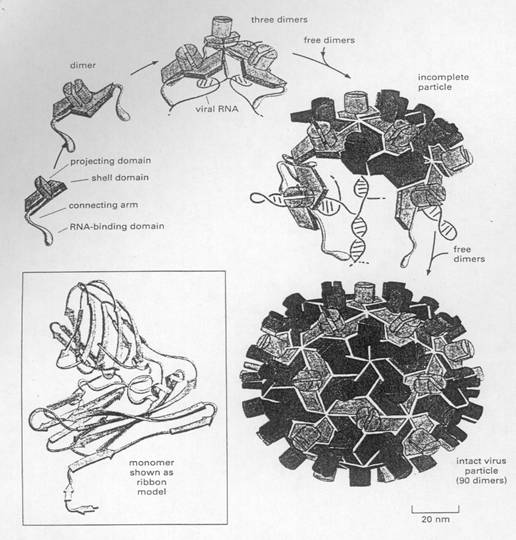
圖27
:
球形病毒的結構。在許多病毒中,相同的protein
subunits會組成一球形的空殼(capsid)以包圍病毒的基因體(genome)。因為幾何學上的因素,不超過60個相同的subunits才能以完全對稱的方式結合在一起。如果些微的不規則能被允許,就能有更多的subunits參與,而能組成更大的capsid。此例為tomato
bushy stunt virus(TBSV),為球形的病毒,具有一個由180個相同copy所形成的巨大capsid。
在細胞內形成許多高分子複合物的資訊一定包含在subunit本身之內,因為純化的subunit在適當的條件下能自動組合成最終結構。第一個能從組成部分自我組合的巨大高分子集合體(macromolecular aggregate)為菸草鑲嵌病毒(TMV)。這個病毒是一個由圓柱狀蛋白環繞一條螺旋的RNA core而成的長桿狀病毒(圖28)。如果分離的RNA與protein subunits一起混合於溶液中,它們便會重新組合成具有完全活性的病毒顆粒。這個組合過程是令人意外的複雜,其中包含了雙環蛋白的形成,雙環蛋白扮演如同中介者的角色,用以幫助病毒外套的形成。
另外一個複合高分子集合體能從組成部分再組合(reassemble)的是細菌的核醣體。其結構由55個不同的蛋白質分子與3個不同的rRNA所構成。如果將分離的這些組成部分培養於適當的條件下,它們便能自動的再形成原本的構造。最重要的是,這樣重建而成的核醣體能夠執行蛋白質合成的工作。正如我們所預期的,核醣體的再組成遵循著一個特殊的途徑:當特定的蛋白質和RNA結合後,這個複合物接著被其他的蛋白質所辨識,如此進行下去直到結構完成。

目前還不清楚更精細的自我組合過程如何被調控。然而另外一個令人好奇的現象是,許多在細胞內的結構似乎擁有一個大於其組成分子(component
macromolecule)好幾倍的的長度。這個特定長度的決定如何達成,仍舊是個謎。目前有三個可能的機制(圖29):最簡單的情況是,一個長的核心蛋白質或其它的的大分子提供一個支架,以決定最後的assembly所能延伸的長度。TMV顆粒便是利用這種方式決定其長度的機制,其核心由RNA所提供。相同的,細肌纖維及其他的細菌病毒的長尾,也是利用一個核心蛋白來決定其長度。
圖28 : 菸草鑲嵌病毒(TMV)的結構。 (A) 病毒顆粒的電顯圖。病毒顆粒包含了一條長的RNA分子被包在一圓柱狀的蛋白質外套(protein coat)裡。Protein coat是由很多相同的protein subunits所組合成的。 (B) TMV的部分結構模擬圖。一條長6000個核苷酸的RNA分子被包在一個螺旋形的外套裡。此外套由2130個相同的coat protein所構成。純化的RNA和蛋白質分子在試管中可以自己組合成具有感染能力的完整病毒顆粒。
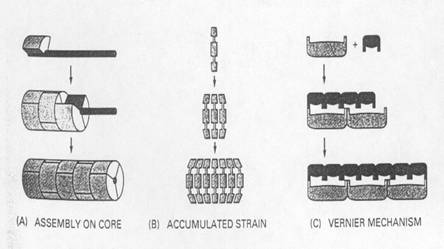
圖29
:
大蛋白質組合(assembly)時決定其長度的三種機制。
(A)
有一瘦長形的核心蛋白或其他巨分子當作測量的依據,沿著它作assembly。
(B)
當polymeric
structure的張力累積到一定程度時assembly就會停止。
(C) vernier type的組合方式。有兩組長度不同的桿狀分子形成錯開的複合物,會一直延伸下去直到兩者的末端吻合為止。
20.複合生物結構(Complex Biological Structures)的形成通常需Assembly Factors的協助
並非所有由非共價鍵結合的細胞結構都能自我組合。粒線體、纖毛或是肌肉細胞的肌纖維就無法在溶液中由其組成分子自動形成。這些例子裡,部分的組合資訊是由特定的酵素及其他執行模板的蛋白所提供,引導構築但並不參與在最終合成的結構裡。
甚至是比較簡單的結構也可能會缺少一些合成必須的構成要素。在某些細菌病毒(bacterial viruses)的形成中,例如頭部,是由許多單一蛋白質次單元在第二種蛋白質構成的臨時鷹架上構成。因為在最終的病毒顆粒第二種蛋白並不存在,所以頭部的結構無法在兩種蛋白質分離的情形自動組合而成。另一個例子是,蛋白質水解在一般的assembly process中是重要且不可回復的。就連某些小蛋白的assembly亦是如此,如結構性蛋白質膠原蛋白(collagen)及賀爾蒙胰島素(圖30)。從這些簡單的例子來看,要組成較複雜的結構像粒線體或線毛結構,也是需要配合其他細胞組成所提供在時間上及空間上的狀況。
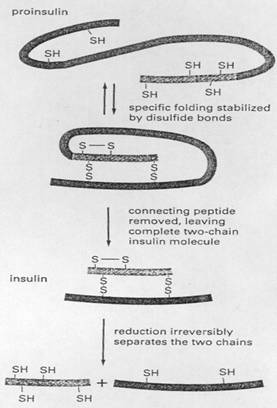
圖30
:
如果胰島素的雙硫鍵被破壞,則無法有效率地再自動重新形成。胰島素一開始會先以proinsulin的方式形成,然後當其摺疊成特定的形狀後,便會被蛋白質水解酵素切掉一部分。被切除的那部份包含了一些資訊是蛋白質自動摺疊成正常構形所需的。所以當胰島素的雙硫鍵被破壞後,其A
chain和B
chain就很難再自動締結合回去。
21.總結
蛋白質分子的三度空間結構是由其胺基酸序列來決定。摺疊的結構則藉由多肽鏈上不同部分之非共價鍵的交互作用而達穩定。擁有疏水性支鏈的胺基酸傾向群聚於分子的內部;而鄰近的peptide bonds間會產生局部的氫鍵交互作用,因而產生αhelix和βsheet的結構。
球狀區域,卽domains,是建構蛋白質的模組化單元(modular units);這些功能區一般包含40至350個胺基酸。典型的小蛋白質,由單一的domain組成;而大的蛋白質則由短的多肽鏈連結數個domains構成。當蛋白質進一步發展,domains會被修飾且和其他domains結合而構築出新的蛋白質。參與很多蛋白質合成的domains稱之為蛋白質模組(module)。至今,在大約一萬個已知的蛋白質結構中,發現一個domain約有1000種不同的摺疊方式。
蛋白質間也利用非共價鍵相互結合而組合成更大的結構。如果蛋白質上具有能與自己表面結合的binding site,則其可組合成二聚體(dimer)、封閉環、球殼、或螺旋聚合體。雖然將蛋白質與核酸在試管中混和,它們能自動組合成複合的結構(complex structure),但有許多的生物組合過程還是牽涉不可回復的步驟。總結的說,並非所有細胞內的結構都能在分離後再自動組合。
22.參考資料
1. Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Robers, Peter Walter. 2002. Molecular biology of the cell, p129-156, 4nd ed. Garland Science, New York