Plasmid display, spatial address
整理:王昌恩
5.ELISA(enzyme-linked immunosorbent assay)
7.Fluorescence cross-correlation spectroscopy (FCS)
Plasmid display利用據附著能力的材質作分析,在這個方法中使用一個DNA附著蛋白和質體DNA間蛋白質和DNA序列的非共價結合,第一個被利用的DNA蛋白是乳糖抑制子,乳糖抑制子由Lac I基因所表現出來,並緊密的結合在特定的DNA序列上lac O,就如同下圖所表示,一個108具有12個胺基酸所組成的。
Peptide library去和乳糖抑制子的C端去做融合,藉著選殖質體pMC5下遊的lacI基因並包括了並排的lacO基因,在細胞內這個質體會直接合成乳糖抑制子和12個胺基酸序列組成的小段peptide,緊接著會形成4隅體的形狀,去附著在質體上的lacO的位置上,在打破細胞後,這一段peptide和質體的複合物利用具有選擇性的免疫抗體將特定的peptide - plasmid複合體給選質出來。
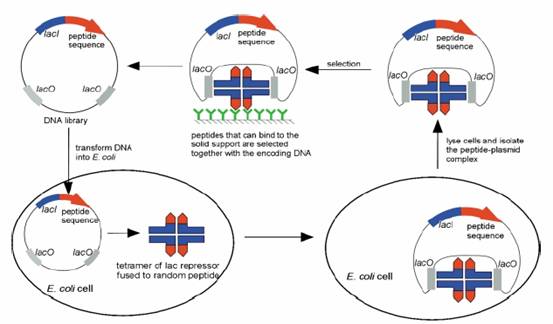
fig.1 Plasmid display
在這一個方法中,peptide會展現多價的形式,在乳糖抑制子中,以4隅體的形式附著在DNA後,每一個質體附著上兩個4隅體,因此這一個方法對於選殖低到中附著力的附著物是非常適合的,Gates等人更發展出一個乳糖操作組headpiece 雙隅體作用DNA附著的區域,這個headpiece 雙隅體可以呈現出較少的peptide library 數目,因此更適合去選殖出高附著力的附著物,儘管DNA附著的在headpiece 雙隅體的機制上不是很清楚,但這一個方法的peptide library會受限於DNA轉型進入細菌內的效率。
1997年Cwirla等人的報告中指出,其成功了分離了一個對thrombopoietin受器的nanomolar peptide的競爭物,依照我們有興趣的觀點來看,作者不僅使用了peptide on plasmid也使用了phage display和 ribosome display,它使用的是VIII的phage display、ribosome display和乳糖抑制子利用各種不同的展示方式,更進一步增加了IC50s由20mM到60nM,然而他使用了更進一步的區間為IC50s由20mM到60nM peptide突變株,然而他使用headpiece 雙隅體方式被授命為高附著力的peptide,AF12505,具有2nm的IC50s,這個AF12505的雙隅體展現出了0.5nm的IC50s,具有可以作為人類重組凝血因子的潛力,儘管回顧者並不清楚,但是這這個作者定論peptide on plasmid選擇上是在生成高附著性peptide附著物最具有效果方法,其應用的重要性是可以利用高價的分子來做peptide的附著,高的價數可以在早期附著一些低附著性的附著物,然而如果已經知道這個附著體是屬於高附著的這樣我們僅需要使用低價數的分子即可以挑選。
Spatially addressable的方法,就是利用在microtiter plate和patch利用固態的固定獨特的address來分辨peptide和蛋白質,所以所有的蛋白質都可以到堆得到原來的DNA序列,我們常常使用microtiter 或是 96-well plate來做分析的工作,近年來更開發出將蛋白質以物理的固態方式固定在平板上,以製成蛋白質晶片。
96-well plate的使用可以歸朔到1950年代,microtiter plates被使用在ELISA(enzyme-linked immunosorbent assay)上面,自從這一個技術在70年代被開發出來之後,ELISA是敏感度很高的一種檢測方式,可以去測量抗體和抗原間的結合,藉著抗原吸附在96-well plat的壁上並和帶有酵素的抗體相結合,在洗去沒有附著的抗體之後去偵測抗體上所附著的那個酵素活性,然後我們使用篩選的方式來篩選蛋白質和peptide library的方式就相似於ELISA,蛋白質和peptide的溶液在分配到每個well,並利用適當的分析方式在每個well中表現。
fig1.微量滴定盤的使用(http://140.112.78.220/~juang/BCT/Chapters/Chap4-2.htm)
5.ELISA(enzyme-linked immunosorbent assay)
酵素連結免疫分析法已朝向普遍化,因為非常靈敏且不必如免疫螢光術和放射免疫分析法需要特殊設備。此法的原理是以一種酵素連結到抗原或抗體上,用酵素活性來做為定量標記。有許多方法已經架構完成,視使用的酵素性質及待測的抗原-抗體系統而定。其中廣泛使用的便是酵素連結免疫吸附分析法enzyme-linked immunosorbent assay,可以測定抗原或抗體。適用於許多病毒的診斷及確認。
ELISA是在1971年首次由Engvall和Perlman所介紹。不論是測抗原或抗體都有很高的敏感性及專一性,且使用設備相較之下少了許多。固相吸附准許unbound reagents快速且完全地清洗。是一種安全又穩定的方法。若要測定抗原,則以一已知特定抗體結到固相之上,含有抗原之待測物加入後,沖洗掉多餘部分,再加入第二種有酵素標記的抗體。此試驗中的抗原至少要有二個決定部位。洗去第二種抗體後,加入基質,用色度計估計酵素活性,此濃度與抗原濃度有關。偵測Ag的方法有兩種:Antibody sandwich immunoassay(即加入Ab再加Ag最後是Ab)及Antibody capture immunoassay(Competition assay)。我們要談的是抗體三明治免疫分析法:首先是Ag專一性的primary Ab的吸附到塑膠盤,此時洞被高濃度的非專一性蛋白所填充(Bovine serum albumin, gelatin, skim milk, or gamma-globulin)。接著加入樣本抗原;那些沒有接合到primary Ab的都被沖到了。最後則是加入有酵素標定的secondary Ab,再洗掉多餘的受標定抗體。留下的受標定抗體被額外的酵素受質所加入,然後我們用1N的H2SO4來終止反應。(上述實驗的沖洗液為Tween 20)。若要測定抗體,則使已知特定抗原固結於某種固相(如塑膠杯或microplate),與待測液血清的稀釋液一起靜置。加進某種特定受質,即可測出酵素活性;變色反應可用色度計(colorimetry)來估計。酵素反應是結合抗原的抗體量的直接函數。包括有Antigen capture immunoasssay和Indirect antibody capture immunoassay.ELISA在臨床診斷學上的應用包括:德國麻疹抗體之測定;CMV,HSV,measles,mumps,VZV等病毒抗體測定。HIV抗體測定(Rapid ELAVIA,SERODIA-HIV)及C型肝炎病毒抗體測定(C型肝炎病毒亦可用PCR來測定)。最後就是判讀結果,在SERODIA-HIV中,紅色顆粒凝聚成一個小紅點沉落盤底,上清液為透明狀即為陰性。若紅色顆粒均勻散布於溶液之中,不會聚集成一紅點,呈混濁狀,即為陽性。若介於中間須重做,再次得同樣結果則要用西方點墨法來做確認。(注1)
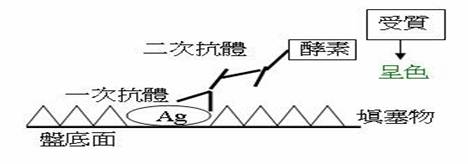
fig.2 ELISA盤底上和抗原抗體結合(http://science.scu.edu.tw/micro/1024/learn/02micro_bio/chao000/chao019.htm)
在Microtiter-Plate Assay中,成千的蛋白質樣本,包括細胞萃取物和純化的蛋白質,必須要一開始先準備,然後轉移到成千個Microtiter-Plate中,然後利用生化的方式在每個well中反應,並去分辨出每個蛋白質的是否具有我們想要的功能,因此一個很主要的挑戰就是快速而花費有效率的純化成千個蛋白質,如果我們想要的活性可以利用很粗糙的分離細胞萃取液就可以被分析,這樣就可以解決了很多技術上的問題,當純化蛋白質的技術被發展出來後,我們可以測活性表現的範圍,可以大量的增加,Martzen等人利用GST融合的蛋白來解決了純化的問題,很特別的,他純化了全部酵母菌6000個ORFs,使用GST標記和選殖ORF library的許多酵素活性,表現在酵母菌(S.cerevisiae)上 ,其中的技巧在如何架構一個GST融合的library,而ORFs利用兩步驟的PCR技術程序以廣用的引子來做放大,利用這個技術來製作將來可用來運用的library, 在這樣的例子中,在一個控制下的ORFs可以誘導一個銅離子誘導的啟動子,這樣的結果在6144個酵母菌俊株中,每一個都誘導含有酵母菌的ORFs去融合GST,生長排列在65 96-well plate中,首先這些酵母菌菌株生長在65 pool在每一個96-well plate作為一個pool,並然這些GST-ORF融合的蛋白質,並純化並排列在64 pools,然而這些正反應的pools在被拿去做同樣的測量在96 well的plate中排列,並去測量其酵素的活性並去分辨GST-ORGs的pool,這蛋白質我們利用tRNA 2’-phosphotransferase和cyclic phosphodiesterase的活性分析法,並使用放射性液態薄層色層分析法,然而用這種方式我們成功選殖出一些先前不知的酵素分子,還有一個重要的功能是Microtiter-Plate提供了一個很有利的工具去連接蛋白質到DNA序列的層次,但是並不能同時提供篩選和選擇的工作,然而,其優勢在很多傳統的生化分析方法上都採舉這一種模式或是自動化模式,最受歡迎的分析方式包括了sintillation proximity assays、紫外光吸收分析法、化學冷光分析法和螢光分析法,螢光分析法具有獨特的功能性,例如FRET和螢光極性的變異性就相當一般化,這個趨勢在Microtiter-Plate分析上體積小,單位密度高的方向來發展,以至於現在的基本孔數已經進展到了384或是1536個well,螢光分析的定律因為具有高敏感度和快數的篩檢,並增加注意力在提升生產力的方式,例如 Eigen和他的同事運用了雙色的螢光雜交關聯性測量儀(fluorescence cross-correlation spectroscopy :FCS)來偵測限制酵素的催化活性,他們將這技術給予命名RAPID FCS(rapid assay processing by integration of dual color FCS)一個 66bp的oligonucleotide使用限制酵素去切的位置使用螢光劑Cy5標示,另一端使用Rhodamine Green標示,交叉分析後可以分辨出哪些是有被酵素切割的,而哪些是沒有的oligonucleotides樣本,大概每個樣本只需要一秒鐘就可以分析出雜交的相關值,這個樣本的體積只需要測量一些microtiter,短的分析時間以及少量的分析體積造成RAPID FCS更適合在大量生產的分析方式中被使用來篩選。
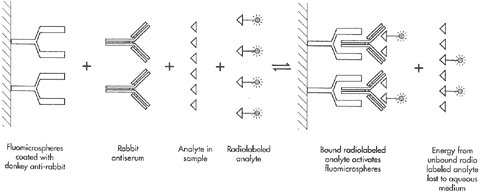
fig3. Scintillation Proximity Assay principle.(http://www.bioprocess.amershambiosciences.com)
7.Fluorescence cross-correlation spectroscopy (FCS)
分子擴散的實驗室測試是最簡單的螢光關聯光譜測量應用,因此常用來校準系統的測量體積。一旦測量體積確定後,這一方法可以測定複雜環境中不同分子的擴散系數。最令人感興趣的是這種方法在活細胞中的應用,例如測量生長在玻璃樣片上的含有加強的強綠色螢光蛋白質(enhanced green fluorescent protein, EGFP)的HEK293細胞。利用雙光螢光成像術得到這些細胞的圖像後,激光可直接照射在感興趣的區域進行螢光相干光譜測量,從而能測定活細胞中不同區域內EGFP的流體運動,細胞內的擴散測量提供了一種對生物體進一步認識的方法。然而在活細胞中應用螢光相干光譜測量時需注意:細胞膜可能會改變測量體積的大小和形狀,細胞的大範圍動態重組也可能嚴重地影響實驗結果,背景螢光同樣會影響相干光譜曲線。精確地測量擴散系數同樣可以用于定量分析特定分子之間的相互作用,此應用可通過對擴散原理的分析或分子計數來完成。擴散分析(diffusion based analysis)和螢光偏振分析 (fluorescence polarization assays)很相似,它們的擴散系數均因兩個不同分子的結合而變化。其主要區別在於偏振分析基於旋轉 (rotational)擴散而螢光進入細胞,細胞調節和控制中均十分重要。在實驗中,每個縮胺酸均被一個螢光染劑分子標定,而importin-a蛋白質無任何標定。importin-a蛋白質的分子重量是60kD,遠大於含有20個胺基酸的縮胺酸分子。因此,接合後的縮胺酸-importin複合體的擴散要遠小於自由縮胺酸的擴散速率。這兩種極端情況,即所有的縮胺酸都和importin-a黏合或兩者間無任何黏合。從這兩鞗曲線中確定該兩種結構的擴散系數后,在固定縮氨酸濃度和變動 importin-a濃度的條件下進行一系列的螢光關聯光譜實驗。在不同importin-a濃度下,縮氨酸分子的黏合百分比可通過擬合的方法獲得,該方法提供了十分精確的蛋白質-蛋白質相互作用的定量表示,而且還有許多其他的應用。(注2)
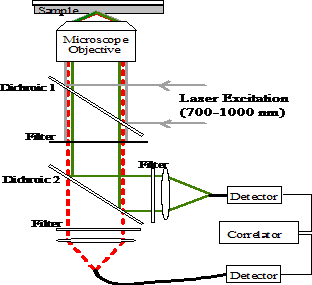
fig.4 fluorescence cross-correlation spectroscopy http://psroc.phys.ntu.edu.tw/bimonth/v24/421.doc
microtiter plates assay可以將每個反應分離的進行,因此非常適合用來測定特殊的酵素活性,Hilvert和他的同事使用96-wellplate分析作為具催化能力抗體在催化能力重組上的第二次的篩選工作,他們篩選了成千個單株抗體來對抗反應中暫時出現的過度相似物,並可以分離一個可以放置代替性benzisoxazoles進入2-cyanophenols的抗體抗體,使其反應速率可以到達108,第一個可以對抗這個反應的中間生成物的單株抗體來自於融合的遺傳工程技術,然而,大約有1000個抗體被轉移到96-well plate中,並可以利用增加340nm吸收光譜來增加2-cyanophenols的形成,以篩選出我們想到得到的酵素活性,其篩選的方式如下圖所表示:
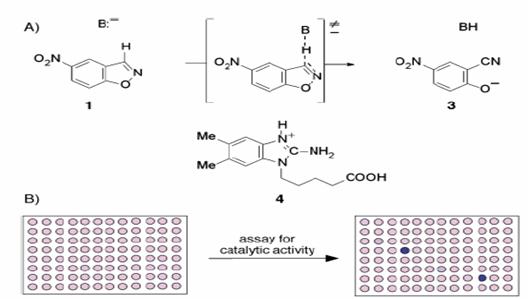
fig.5 High efficiency catalytic antibodies generated by using transition state analogue and microtiter plate screening.
這些抗體中的兩個被我們選來做進一步的分析並展示出3.4*108和3.5*107速率加速度,並與未催化的反應做比較,如下表:
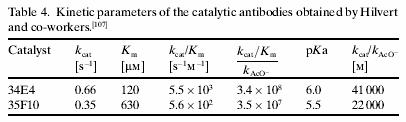
傳統上,僅有10-100個我們想要活性的單株抗體可以被選出,並且在每一個抗體中我們分別以酵素動能來做分析,這樣本所得到的108倍的反應加速度遠比正常得到的催化活性的抗體和大規模直接催化篩選的活性和利益來的大的多。
microtiter plates assay的普遍使用,不僅僅是在抗體的工程上,更包括了一般性的蛋白質工程,例如Giver 等人篩選出突變的枯草桿菌Bacillus subtilis p-nitrobenzyl esterase (pNBE)library去解決了pNBE可以適應溫度變化而仍具有催化功能,他們將這個蛋白質利用PCR做點突變並加入一段DNA片段並送入大腸桿菌中表現,正如下圖所示:
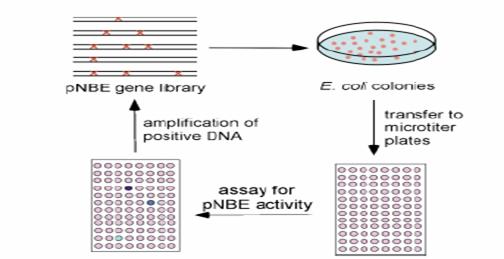
fig.6 The evolution of thermostable pNBE.
利用這種方式可以生成500-2000個菌落,在將每一帶挑選放置於96-well plate中,pNBE的活性可以被我們所測量分析,並利用p-nitrobenzyl acetate的分析方法來分析細胞脆取物中chromogenic產物的生成量以及活性,經過這樣的點突變處理後,我們篩選穩定化且有意義的pNBE並提高他的Tm 14˚C,令人覺得有趣的是,這個突變過後的酵素並會不在室溫中有所改變,這樣的運用性可以非常的成功,因為這樣的一個技術可以增加了以知蛋白質溫度的可變性,由於我們可以利用這樣方式來測量esterase的活性,所以我們更可以藉此技術來做膜蛋白library的挑選,Spatially addressable library似乎是將每個樣本都採以獨立的空間去做篩選的動作,可是適合用來篩選cDNA的library,他需要較少的蛋白就可以來做測定,並可以來檢測其表現的活性量,Haushalter利用活體化的選殖來分辨出DNA的糖化作用從一個非洲牛蛙的cDNA資料庫中去做篩選出來,這樣的cDNA資料庫可以分作120個pools,每一個大約包含了100 組cDNA,這樣的cDNA pool可以再活體外microtiter plates的well中作轉錄和轉譯反應,我們在利用放射線標定,作為在轉錄作用合成的蛋白質的glycosylase的抑制劑,使得更易於去跑native 的polyacrylamide電泳,這樣的pool可以利用區隔的空間來分別出單一的clone對抑制劑附著的區別性,並可以分別出單體的不同活性,這樣的模式可以用來做單一股選擇單功能的uracil-DNA糖化作用的篩選。
儘管基因體序列解碼帶給基礎生物醫學研究有很大的進展,然而有了基因序列資訊卻無法完全掌握其表現出來的蛋白質所應具備的分子功能與結構,並存在細胞中的位置以及扮演特定生物調控與反應路徑,甚至跟致病機轉的關係為何,還尚未得到有意義的解答。因此,生物學家需發展高產量實驗技術(high-throughput experiment),用來同時測量成千上萬的基因表現(gene expression),所以生物晶片發展就非常重要。 最早所研發的生物晶片為基因晶片或基因微陣列(DNA chip or DNA Microarray),其偵測是細胞中mRNA濃度或量的變化程度。可是,真正影響細胞的生理狀態是基因產物-蛋白質,然而我們藉著已經開發出來的DNA chip所得到的經驗,更進一步去開發蛋白質晶片。有別於RNA層次,蛋白質需經過轉譯後修飾(post-translational modification)及任務分派(sorting)。如此多樣化的蛋白質生成合成資訊,加上功能性蛋白質間或蛋白質與其他小分子/藥物/巨分子間的交互作用(interactions),縱使利用比較傳統的方法(如ELISA,..)都無法同時測量整個細胞中所有蛋白質的變化情形,使得蛋白質晶片(protein microarray)的發展更形重要(注3),然而生醫晶片是功能性生物材料與微機電技術之整合。微機電技術可預期提供晶片之微細規劃設計,而能在小面積範圍內提供多數個微細區域作為功能性生物材料之反應場所。反應過程發生之物理性或化學性訊號,再配合適當檢測系統進行定性或定量。而抗體是對其對應抗原具有極佳專一性親和力之功能性生物材料,尤其單源抗體更只針對特定抗原之特定抗原決定基具備專一性親和力,能夠在成分極複雜之樣品中對其對應抗原進行結合反應而不受其他成分影響。生物晶片、單源抗體、以及適當檢測系統之有效組合,可預期構成以抗體-抗原反應為基礎之多功能腫瘤標記生物醫學晶片檢測系統。
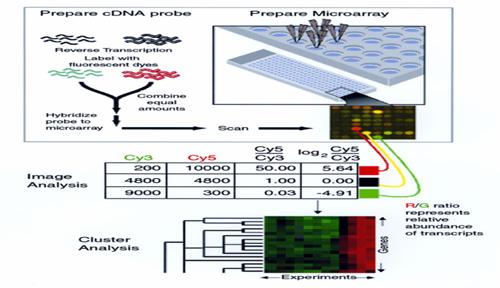
fig.7 http://www.cdc.gov/ncidod/eid/vol6no5/cummings1G.htm
基因晶片與蛋白質晶片之主體在於基材材料以及點製於上的物質;目前發展中之基材材質有玻璃、尼龍膜、硝化纖維膜、矽膠、塑膠與陶瓷等;而點製在基材上的物質當為基因或是寡核 酸或是其類似物時則稱作基因晶片,點製在基材上之物質為蛋白質、 或是多 或是其類似物時則稱作蛋白質晶片。當檢測樣本中含有可與基材上點製物質結合之對應物質時便會在檢測之時互相結合,並藉由連結可偵測之標示物質而可加以偵測。生物感測晶片係為一整合型晶片,組合了酵素、配位體與受體、抗體與抗原、合成胜鏈與核酸等生物分子識別材料與物理、化學等可交換能量元件,使之成為可測定微量成分的生物感測裝置。實驗室晶片又稱為微處理程序型晶片,運用微流體與表面化學技術,利用微小化結構可以將實驗室之實驗流程步驟微縮至基質上而可將樣品製備、生化反應與結果檢測一氣呵成。像是利用微機電技術以微量PCR與微量毛細管電泳技術作出之電泳及複製晶片(PCR-CE Chip)便可將核酸增量反應及核酸分離與分析完全微縮在晶片上完成。 基因晶片與蛋白質晶片目前主要有三種點製物質之方法,一種是原位合成法,即在基材材料上之預定位置以光引導的方式讓寡核甘酸與寡太逐步合成在基材上;此方法以Affymetrix公司為代表。第二種為合成點樣法,是在他處合成完欲點製物質後才以微量點樣裝置將該物質直接點在基材上;此方法以Incyte Pharmaceutical公司為代表。第三種方法為壓電式打印法,屬於原位合成法之一種,但是以類似彩色噴墨打印的方式將欲合成之寡核苷酸或寡肽原料噴射在基材上並逐步合成寡核苷酸或寡肽。(注4)
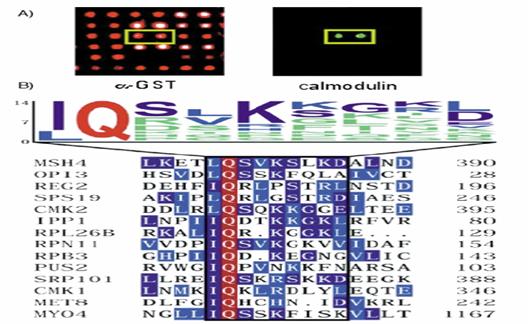
fig8. Assay for camodulin-binding proteins on proteome chips.[
spatially addressable定律的優點似乎能夠適合蛋白質的功能的傳統偵測方式,部分的應用可以在蛋白質的純化上,spatially addressable library被限制在大約104個差異個體的蛋白質,儘管有些的應用是在生化工程上,spatially addressable的方法是一種極佳的適合方式是測cDNA,甚至可以決定蛋白質的功能,如同其他的活體外的實驗,這些檢測的方式也限於蛋白質在活體外要能穩定的存在,此外我們上不清楚利用固態的方式來固定蛋白質是不是會影響蛋白質的功能,我們利用了這些廣泛的化學反應來做傳統反應的檢測,蛋白質被固定在microtiters,可以適合去附著一些蛋白質以及進行酵素催化,這個描述spatially addressable的方式,在技術上所需要的純化和抑制蛋白質都可以利用這種模式來完成,但是在蛋白質晶片上,由於無法同時達到同一時間的檢測,因此我們還是被需將其回推到DNA 的層級,來達到真正的檢測。比起DNA晶片整個蛋白質晶片製造過程所應用的技術相當複雜,這裡就幾個關鍵的步驟及其技術應用原理做一般性的介紹,可將提到的內容做個總結。事實上,蛋白質晶片發展過程乃依循DNA晶片的技術基礎與經驗過程,然而其特有的技術困難將會延遲大量量產反倒是廣泛運用在生物醫學研究領域。
Hening Lin(2002) Screening and Selection Methods for Large-Scale Analysis
of Protein Function , Angew. Chem. Int. Ed, 41, 4402 - 4425
Hening Lin(2002) Screening and Selection Methods for Large-Scale Analysis
of Protein Function , Angew. Chem. Int. Ed, 41, 4402 – 4425
注1:http://members.tripod.com/~talung/ELISA.html
注2:http://psroc.phys.ntu.edu.tw/bimonth/v24/421.doc
注3:http://binfo.ym.edu.tw/styang/seminar/proarray/array_farbication.htm
fig1. http://140.112.78.220/~juang/BCT/Chapters/Chap4-2.htm
fig.2 http://science.scu.edu.tw/micro/1024/learn/02micro_bio/chao000/chao019.htm
fig.3 http://www.bioprocess.amershambiosciences.com
fig4. http://psroc.phys.ntu.edu.tw/bimonth/v24/421.doc
fig.7 http://www.cdc.gov/ncidod/eid/vol6no5/cummings1G.htm
fig.5,6,8 Hening Lin(2002) Screening and Selection Methods for Large-Scale Analysis
of Protein Function , Angew. Chem. Int. Ed, 41, 4402 – 4425